结直肠癌多标记转录分类反映肿瘤细胞群体的异质性
背景:转录分类已被用于将结直肠癌(CRC)分成具有不同生物学和临床特征的分子亚型。然而,目前尚不清楚这些亚型是否代表着离散的、互斥的实体,还是具有潜在重叠的分子/表型状态。因此,我们将重点放在CRC固有亚型(CRIS)分类器上,并评估将多个CRIS亚型分配给同一样本是否提供额外的临床和生物学相关信息。
方法:我们使用CRIS分类器的多标签版本(multiCRIS)对新生成的606个CRC患者来源的异种移植物(PDXS)的RNA测序数据进行了分析,同时结合了人类CRC批量和单细胞RNA测序数据集。比较了单标签和多标签CRIS的生物学和临床相关性。最后,开发了基于机器学习的多标签CRIS预测器(ML2CRIS)用于单个样本的分类。
结果:令人惊讶的是,约一半的CRC病例可以明显地分配给多个CRIS亚型。单细胞RNA测序分析揭示,多个CRIS成员身份可能是由于同时存在不同CRIS类别的细胞,或者较少情况下由于具有混合表型的细胞。发现多标签分配可以改善对CRC预后和治疗反应的预测。最后,ML2CRIS分类器在单个样本分类的情境下被验证具有相同的生物学和临床相关性。
结论:这些结果表明,即使同时分配给同一CRC样本,CRIS亚型仍保留其生物学和临床特征。这种方法有潜力在其他癌症类型和分类系统中推广应用。
该研究于2023年5月发表发表在《Genome medicine》,IF:15.266。
技术路线:
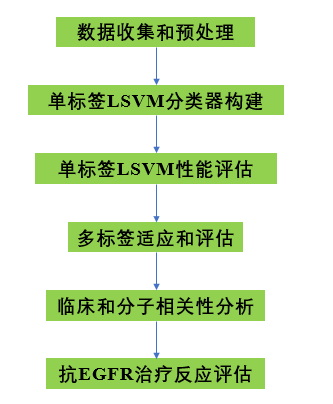
实验方法:异种移植物收集、TCGA和PDX RNA-SEQ数据预处理、CRC单细胞数据及其预处理、bulk/scRNA-seq数据和预处理、scRNA-seq数据的伪批量、CRIS分类、单标签分级机、多标号单样分级机。
1、结直肠癌内在亚型的多标签CRIS分层研究
为了改善结直肠癌的分层,并根据CRIS分类捕捉生物特征,我们推断其最近模板预测(NTP)算法不仅可以用于指定最显著的单一类别,还可以评估每个样本对所有CRIS类别的分配,以及每个分配的虚假发现率。因此,我们实施了基于NTP的CRIS分类器的新的多标签版本,名为“multiCRIS”,能够根据与每个CRIS中心点的距离和其显著性将每个样本分配给一个或多个CRIS类别。
首先,将MultiCRIS应用于来自癌症基因组图谱(TCGA)的620个样本的RNA测序数据集,以明确地将91%的样本至少分配给一个类别(图1a)。有趣的是,52%的样本还可以被确信地分配给其他CRIS亚型(图1b)。
值得注意的是,对于所有的CRIS亚型,次要分配的数量与主要分配大致相等(图1c)。多重分配主要发生在两个特定的亚家族之间:CRISA/CRIS-B和CRIS-C/CRIS-D/CRIS-E。最后,为了评估这些多重分配是否捕捉到具有多个CRIS生物特征的肿瘤,我们探索了与每个CRIS类别相关的主要特征。
有趣的是,分配给次要类别的样本在图1d中显示了类别的关键分子特征,包括CRIS-A中的MSI状态,CRIS-C中的KRAS突变的消失,以及CRIS-D/CRIS-E中的WNT信号通路活性和CRIS-B样本中的上皮间质转化(EMT)。值得注意的是,我们观察到具有多个分配的样本倾向于与CRIS中心点之间的距离较大,这可能反映了同时具有不同表型的细胞组成或具有不同表型的细胞混合的情况。

图1展示了针对TCGA数据集中的596个结直肠癌样本进行的多标签CRIS分类的结果。
2、多个CRIS分配中的单细胞异质性。
观察到一部分结直肠癌的多个类别分配可以通过两种方式解释:肿瘤由具有模糊表型的癌细胞组成,或者存在混合的不同亚型细胞群体。为了探索支持多个CRIS分配的异质性,我们在一个由PDXS(患者源性异种移植)衍生的5个结直肠癌器官样本集合中进行了一系列的配对单细胞RNA测序(scRNA-seq)和批量谱分析。这些数据允许直接比较单细胞和批量转录组谱分析结果。作为第三个选择,通过聚合一个样本中所有单细胞谱分析结果来获得伪批量谱分析结果。值得注意的是,尽管来自单个细胞的谱分析结果平均捕获了至少5个支持读数的1116个转录本,但伪批量谱分析结果平均涵盖了超过17,095个转录本。如预期的那样,匹配的批量/伪批量样本的谱分析结果显示了强烈的相关性,而无法通过非匹配比较获得。这些结果表明,(i)单细胞谱分析结果显示出高度的异质性,以及(ii)聚合的单细胞谱分析结果能够重现批量谱分析结果中所获得的转录组谱。因此,这种3D体外器官样本培养系统捕获了具有复杂转录组异质性的细胞谱。
值得注意的是,我们发现存在同时存在的细胞混合物,每个混合物具有一个单一的CRIS分配,以及具有混合多个CRIS亚型的细胞。来自给定器官样本的个别细胞主要被分配到该器官样本的批量谱分析结果所定义的CRIS亚型/亚型组(图2)。
这些结果强调了在单细胞分辨率下,大多数细胞被分配到单个CRIS亚型,并且它们的混合导致了批量转录组的多亚型分配;然而,也有可能存在一小部分具有混合表型的细胞,在给定的批量样本中对多个CRIS亚型的分配产生贡献。事实上,在所有接受多个CRIS批量分配的器官样本中,我们检测到了具有不同CRIS标识的细胞和具有混合表型的细胞的共存(图2)。
为了将我们的观察扩展到人类肿瘤,我们利用来自一组患者的公共单细胞RNA测序数据(GSE132465),重点关注上皮细胞,比较伪批量和单细胞的多标签CRIS分配情况:这种分析证实了存在多个CRIS分配的患者。在这些样本中,我们证实大多数单个细胞被分配到特定的CRIS亚型(64%的分类细胞,其中75%被分配到单个CRIS亚型,25%被分配到多个CRIS组;图3a)。然而,类似于器官样本,每个样本由不同的细胞群体组成,这些细胞群体被分到不同的CRIS亚型中,导致了一个复杂的表型,该表型通过伪批量分析的多个CRIS分配被捕捉到(图3b)。因此,被分配到单个CRIS亚型的样本往往具有更高比例的被分配到该亚型的细胞。在特定样本中,具有多标签分配的单个细胞的高百分比可能反映出组织中正在经历功能转变或稳定的中间分化阶段。例如,在患者SMC17中发生了这种情况(图3b),其中57%的分类细胞显示出多标签表型。类似地,SMCO3和SMC21患者分别显示出34%和28%的具有混合表型的细胞(图3b),与它们在批量分析中追踪到的多标签状态一致。
总的来说,这些结果表明,CRIS转录组的异质性根源于单个细胞水平,而单个细胞的表型总结起来定义了肿瘤批量的CRIS分类。因此,多CRIS肿瘤的证据主要可以通过具有特定功能特征的不同细胞群体的镶嵌组成或具有混合表型的少量混合细胞来解释。
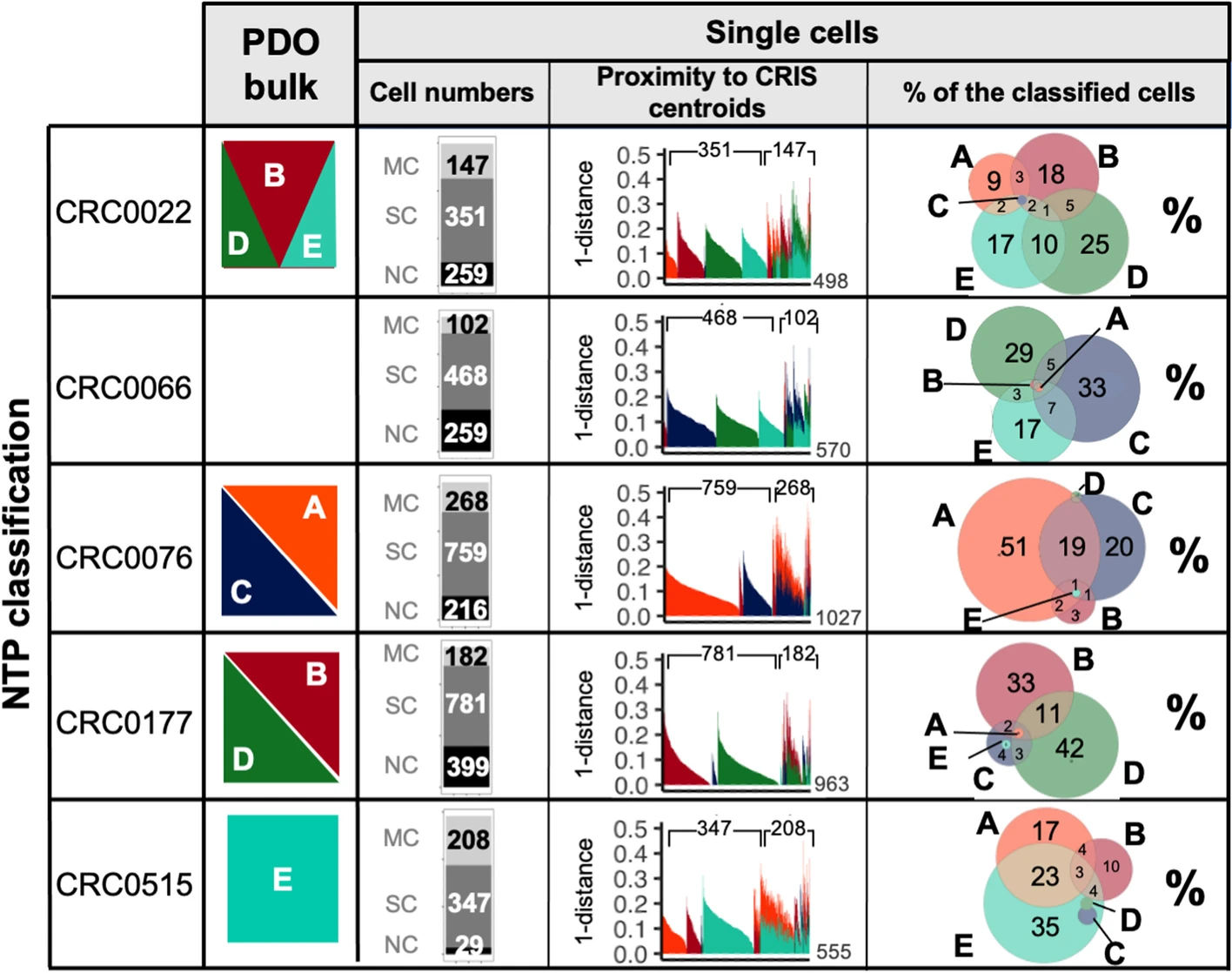
图2展示了人类器官样品的MultiCRIS分类结果。

图3展示了人类结肠直肠癌(CRC)的MultiCRIS分类结果。
3、针对CRIS分类的单样本方法
MultiCRIS为复杂的生物学和临床结果铺平了道路。然而,它受到其NTP实现的影响,该实现依赖于在样本批次上计算的质心距离和基因级别的Z分数,不允许单个样本的分类。为了克服这个问题,我们转向单样本算法,能够独立地对每个样本进行分类:这些算法既可以处理只对主要类别进行单标签分配的情况(SC),也可以处理多标签分配的情况(MC),以捕捉内部的异质性。我们的工作流程如图4所示;它包括对所有算法进行初始训练阶段(蓝色),在测试数据上对它们的性能进行评估(粉色),以及对最有希望的单样本方法进行最终的临床和生物验证(绿色)。我们首先实现了单样本单标签算法,能够识别每个样本的最显著(主要)类别。考虑的方法包括随机森林(RF)、线性支持向量机(LSVM)、多项式(PSVM)和高斯径向基函数(GRBF-SVM)核的支持向量机、神经网络(NN)和极端梯度提升树(XGBoost)。随后,通过提取每个样本的所有CRIS类成员关系,TESE单样本算法被适应于多标签上下文;这使得能够对照多标签NTP(多CRIS)的结果来验证其结果。
为了评估每个算法并确定最适合在临床应用的单样本分类器中预测CRIS类别成员资格的算法,我们利用了来自TCGA项目的原发性结直肠癌样本集(n=562)和来自患者衍生的异种移植瘤(PDXS,n=550)的队列。TCGA数据被分成训练集和测试集,保持整个数据集中CRIS类别比例不变。每个分类器仅考虑CRIS基因的表达值作为特征空间,并使用NTP主类作为目标参考进行训练,无论其是单标签还是多标签的使用。TCGA样本的30%和完整的PDXS数据集被用作两个独立的测试集,以评估单标签和多标签分类器的结果。
在针对主要类别分配的单标签评估中,我们使用全局准确率、精确度和召回率,衍生的度量指标(F1分数和马修斯相关系数(MCC)),以及基于阈值的度量指标(接收器操作特性曲线下面积和精确率-召回率曲线下面积),以评估所考虑算法的性能,并将其与原始的NTP方法的性能进行比较。LSVM在TCGA测试集上达到了约80%的准确率,在PDXS上达到了75%的准确率,在考虑了所有类别的精确度和召回率时表现良好(图5)。虽然XGBoost和RF取得了有趣的表现,但它们的类别特定行为似乎不太稳定,并且整体上略逊于LSVM(图5)。
此外,在单标签情况下,机器学习方法与TSP方法进行了比较,TSP方法是单样本CRIS分类器的首次尝试,最初显示出与NTP分类的相当有限的一致性。本研究中证实了其次优结果:我们所有的分类器都比TSP获得了更高的准确性,最高准确率为70.7%。特别是对于LSVM来说,即使在TCGA测试集上F1分数最低的类别(CRIS-E,为73%)也显著超过了TSP的结果(CRIS-E为28%)。
在PDXS数据集上,LSVM取得了最令人信服的结果。在TCGA中,CRIS-A是最常见的类别,略多于CRIS-C;其次是CRIS-E,而CRIS-B和CRIS-D的规模较小但可比较。在PDXS中几乎可以观察到相同的趋势,例外就是CRIS-A类在PDXS中的表示较少,这是因为来自转移性结直肠癌的样本中MSI病例(CRIS-A富集)的稀缺性。
因此,基于性能评估,我们确定LSVM作为最佳的单标签分类器,用于预测每个单个CRC样本的主要CRIS类别。然而,所有三种训练过的算法都能通过算法自适应技术计算出对所有5个CRIS类别的隶属度,为多标签情景奠定了基础。

图4展示了基于机器学习构建单样本CRIS分类器的工作流程。

图5展示了基于机器学习的CRIS分类器的性能评估结果
4、通过单样本方法,进行多标签CRIS分类。
在算法自适应策略的基础上,我们开发了多标签适应(mla)的单样本CRIS分类器。具体来说,每个mla算法从其单标签版本继承了主要类别的分配能力,但可以将任何异质样本与一个或多个额外的次要类别关联起来。
为了评估mla分类器,我们使用了与单标签类似但适应于多标签环境的度量指标(放宽准确率、精确率、召回率),以及特定的多标签度量指标(平均精确率、Hamming损失、子集准确率和多标签准确率)。所有这些指标都将mla算法的结果与本研究中引入的MultiCRIS方法获得的目标分配进行比较。
在mla算法中,LSVM在考虑类别精确率和召回率时仍然达到了最佳整体性能,显示出在多标签环境中也是最稳健的方法(图5)。此外,LSVM在91.7%的情况下分配了主要的多标签CRIS类别(即根据NTP算法确定的最突出的类别),并且在预测TCGA测试样本的多标签特征时达到了92.6%的平均精确度。在考虑Hamming损失时,即错误分类标签的平均比例,LSVM在TCGA测试集和PDXS集中都具有最低的损失比例。最后,LSVM的子集准确率(严格相同的标签归属)也非常重要,尤其考虑到每个算法只通过提供主要类别作为参考目标进行训练。
因此,LSVM显然是在单标签或多标签视角下执行单样本分类的最佳方法。
5、单标签和多标签LSVM分类器的临床和生物学评估
我们首先评估了基于LSVM模型在TCGA数据集上的预后价值,考虑了不同的情景:仅考虑作为主要类别分配的样本、仅考虑作为次要类别分配的样本,或者考虑所有被分类为CRIS类别的肿瘤,不论其是主要还是次要分配。在所有这些情况下,使用Fisher检验进行的任何比较都是针对那些完全未被分配到所研究的CRIS类别的样本。对于基于NTP和LSVM的单标签分类器,Kaplan-Meier(KM)生存分析证实了CRIS-B类与不良预后的显著关联(图6a、b)。有趣的是,通过多标签分配,将次要CRIS-B分配的样本从非CRIS-B组中排除,突出了与不良预后的更高的相关性(图6c、d)。因此,当排除具有主要CRIS-B分配的样本进行分析时,次要CRIS-B分配的样本显示出更差的预后(图6e、f)。当将主要和次要CRIS-B病例合并时,预后显著性达到最大值(图6g、h)。值得注意的是,在所有情况下,基于LSVM的分类器具有更高的预后显著性。
在PDXS队列中评估了对抗EGFR治疗的反应。我们确认了CRIS-C与Cetuximab的敏感性相关联,其中单标签LSVM(优势比(O.R.)= 3.281,置信区间(CI)= 1.66-6.73)和多标签LSVM,包括在CRIS-C队列中的次要分配,显示出类似的表现(O.R. = 3.36,CI = 1.24-10.64)。
所有这些证据都证实了我们的LSVM单样本模型的预测结果的可靠性,特别是mla LSVM,即ML2 CRIS(多标签机器学习CRIS)。ML2 CRIS能够突出样本的生物学内在异质性(如果有的话),同时在临床应用中对每个患者进行个体化评估。这证明了ML2 CRIS在临床使用环境中的可靠性。
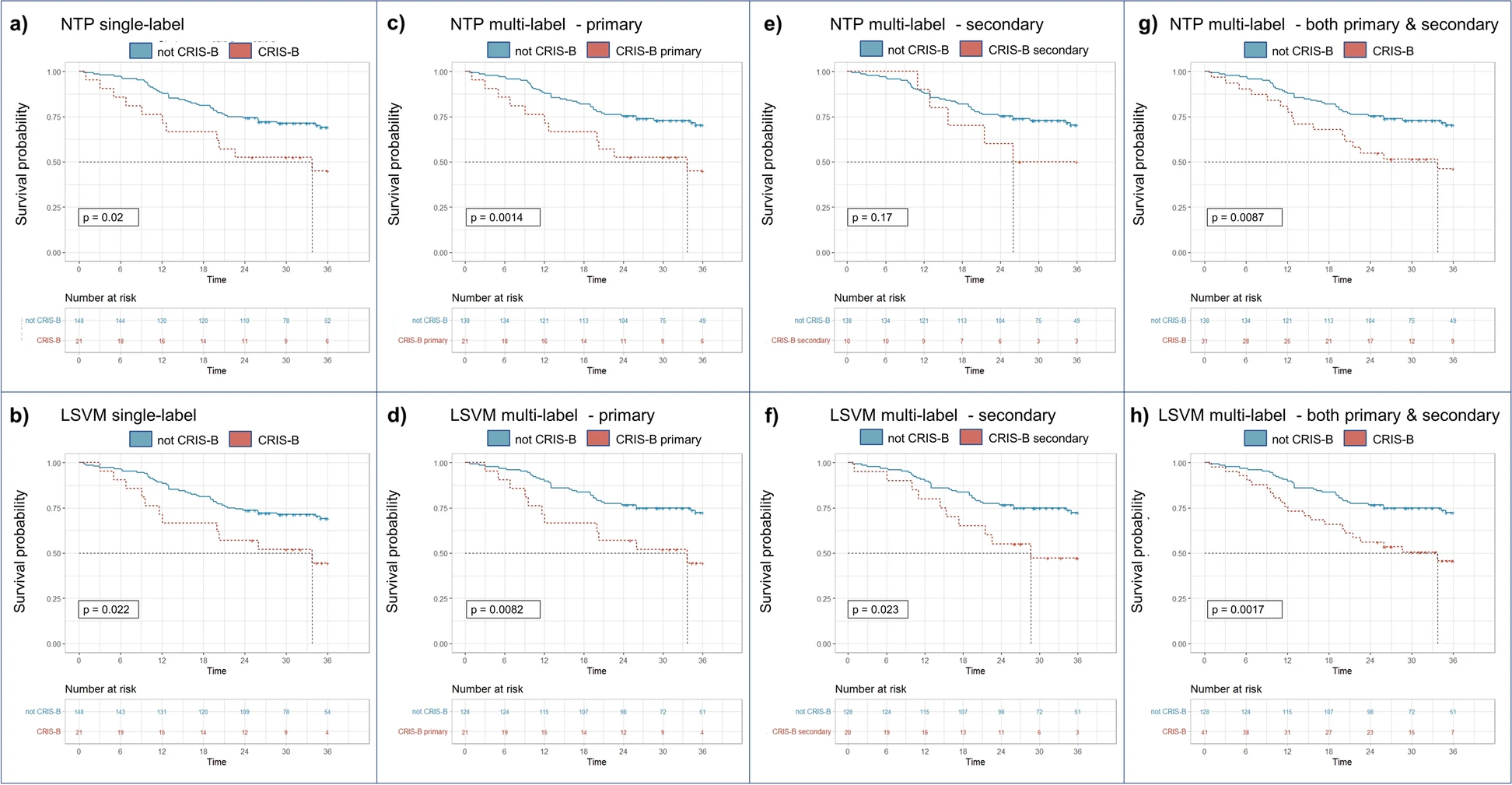
图6展示了单标签和多标签CRIS-B分类在预后方面的意义
参考文献:
Cascianelli, S., Barbera, C., Ulla, A.A. et al. Multi-label transcriptional classification of colorectal cancer reflects tumor cell population heterogeneity. Genome Med 15, 37 (2023).https://doi.org/10.1186/s13073-023-01176-5