mSWISNF复合物在TKIs耐药EGFR突变肺癌中的调控机制

EGFR突变型肺腺癌可获得酪氨酸激酶抑制剂(TKI)耐药,比如奥西替尼,这通常是由非遗传机制引起。作者鉴定了奥西替尼敏感和耐药的EGFR突变细胞和患者肿瘤衍生模型中的染色质可及性变化和基因调控特征,并揭示了哺乳动物SWI/SNF染色质重塑复合物在TKI耐药性中的作用。通过分析mSWI/SNF全基因组定位,作者确定了奥西替尼抗性状态下癌细胞特异性基因靶点。重要的是,基因层面和药理学层面破坏SMARCA4/SMARCA2 mSWI/SNF ATP酶,可通过抑制mSWI/SNF介导的细胞程序调节细胞增殖、上皮-间充质转化、上皮细胞分化和NRF2信号传导,使一些耐药模型对奥西替尼重新敏感。这些数据强调了mSWI/SNF复合物在TKI耐药性中的作用,并表明mSWI/SNF抑制剂在TKI耐药性肺癌中的潜在效果。该研究于2023年8月发表在 《Cancer Cell》,IF:50.3。
技术路线

主要研究结果
1. 奥西替尼耐药的EGFR突变癌细胞发生染色质可及性变化
为研究EGFR突变癌细胞奥西替尼的耐药性机制,作者产生了五对亲代和奥系替尼耐药性的EGFR突变癌细胞系(图1A)。亲本细胞系对奥西替尼的EC50为10 nM或更低,而其抗性细胞系EC50高出>90倍(图1B和1C)。然后对这些细胞系对进行RNA-seq,确定抗性细胞的基因调控谱。作者鉴定到了不同的上调和下调基因,其中许多差异基因在至少2个细胞系之间共有(图1D)。C3和C8分别包含五种奥西替尼抗性而非吉非替尼抗性状态中的下调和上调基因,包括MMP2和FGF1(下调)以及ZEB2、ATF3、ETS-1和FYN(上调)等基因。作者在一个抗性细胞系中鉴定到许多独特的差异调节基因,强调了抗性状态的异质性(图1D)。EMT和炎症反应等通路在某些细胞系中上调,而MYC靶点、干扰素α/γ反应信号通路则显示下调(图1E)。
接下来,作者在四个细胞系对进行ATAC-seq,其中包括PC9*、HCC406*、HCC827*和PC9(图1F)。作者鉴定到抗性细胞中抗性相关的可及性基因组差异位点(图1F)。将这些数据与RNA-seq数据结合,发现>50%基因的表达变化与基因启动子或增强子处或附近的DNA可及性变化一致(图1G、1H)。这些发现证实EGFR突变NSCLC癌症细胞系中抗性和亲代状态之间的染色质可及性变化是表征TKI抗性基因调控程序的基础。

图1:EGFR突变肺癌细胞中奥西替尼耐药的染色质可及性和基因调控基础
2. mSWI/SNF复合物是奥西替尼耐药基因程序和染色质可及性的上游调节因子
染色质可及性变化是TKI抗性的一个关键基因调控特征,接下来作者试图预测控制这些变化的潜在染色质相关调控因子。在所有细胞系中,对奥西替尼抗性(OR)(或吉非替尼抗性)状态下的差异基因表达谱(RNA-seq)进行IPA分析,mSWI/SNF复合物的ATP酶亚基SMARCA4关联度最高(图2A)。其他包转录因子(TF)括,如TP63、STAT3和SOX2,其中一些通过mSWI/SNF复合物以及ARID1A与染色质相关连,ARID1A是典型BAF(cBAF)mSWI/SFF亚复合物亚基(图2A)。
为了解SMARCA4如何调节基因表达,作者使用CUT&RUN分析PC9*/OR*、HCC406/OR和HCC827*/GR6细胞系中mSWI/SNF复合物全基因组的占有情况(图2B)。将这些数据与ATAC-seq进行整合,确定了在抗性状态下获得或丢失的位点子集(图2C)。获得和丢失位点主要位于启动子远端,并且在AP1家族基序上富集(图2B、2C)。对这些基因进行排序,其在抗性状态下mSWI/SNF特异性结合与可及性变化表现一致(图2D)。EMT(TWIST1和ZEB2)、细胞迁移(MMP13、BMP4和COL4A1)和RTK信号传导基因上调,而编码上皮细胞分化和信号传导(FGFR1/2、JAG1和NOTCH1)的基因在抗性状态下下调。参与MAPK信号传导的基因表达(MAPK1、DUSP6和SPRY4)以细胞系特异性的方式发生改变(图2D)。亲本和抗性状态之间mSWI/SNF占有率、可及性和基因表达的一致变化发生在ETV1和TWIST1基因座(图2E)。这些发现表明,在TKI抗性EGFR突变细胞系中,mSWI/SNF复合物在抗性相关基因座上的活性起着关键作用。
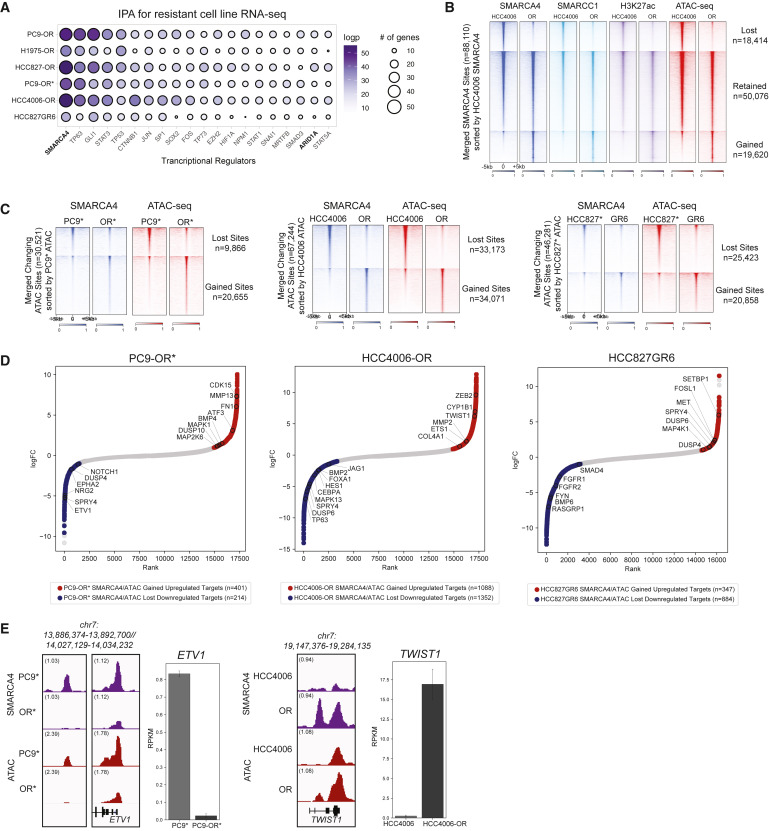
图2:哺乳动物SWI/SNF(BAF)复合物是抗性相关基因的关键调控因子
3.抑制SMARCA4使奥西替尼耐药模型对奥西替尼重新敏感
作者在PC9/PC9-OR、H1975/H1975-OR和HCC827/HCC827-OR亲本和抗性细胞系中进行shRNA介导的SMARCA4敲除(图3A-3D)。与H1975-OR和HCC827-OR细胞相比,SMARCA4敲低对奥西替尼处理后的PC9-OR细胞影响最显著。与未处理细胞相比,奥西替尼的存在使PC9-OR细胞增殖完全消除(图3B和3C)。此外, SMARCA4敲低后奥西替尼EC50降低了8倍(图3D)。
同时,作者利用对奥西替尼具有敏感性的EGFR突变肺癌的患者衍生异种移植物(PDX)来观察SMARCA4敲低的影响。通过免疫组织化学(IHC)检查这些PDX中SMARCA4的水平,发现所有肿瘤都产生SMARCA4(图3E)。因为YU-005X对奥西替尼表现出原发性耐药性,作者使用源自该肿瘤的细胞系(YU-005C细胞)。SMARCA4敲除显著损害YU-005C细胞增殖和形成集落的能力,而添加奥西替尼则加剧了这种效果(图3F和3G)。体内实验中,作者在YU-005C细胞来源的肿瘤中敲除SMARCA4(图3H和3L)。皮下注射YU-005C细胞导致肿瘤形成,SMARCA4基因敲除与多西环素处理后肿瘤继续生长(图3H)。SMARCA4基因敲除和处理则抑制肿瘤(图3H和3I),而对照SMARCA4野生型肿瘤生长没有受到奥西替尼处理的影响。这些结果表明,奥西替尼耐药肿瘤在奥西替尼存在下依赖SMARCA4进行增殖。
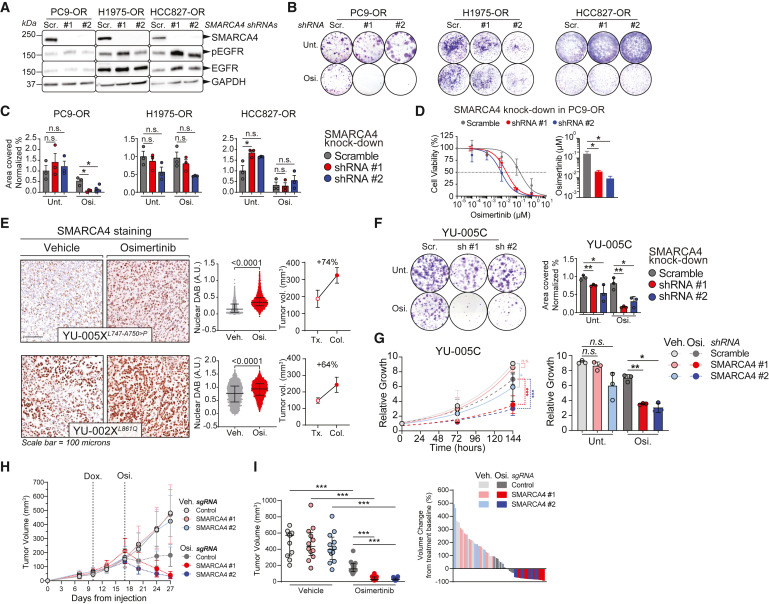
图3: SMARCA4缺失使一部分耐药肿瘤对奥西替尼敏感
4. SMARCA4丢失改变染色质可及性、抗性和增殖相关基因谱表达
为研究SMARCA4如何在奥西替尼存在的情况下维持癌症细胞,作者在PC9-OR和YU-005C细胞中在奥西替尼存在或不存在的条件下敲低SMARCA4,然后进行RNA-seq和染色质可及性(ATAC-seq)分析(图4A)。两种细胞中均检测到上调和下调基因,与对照相比,并确定了奥西替尼处理后SMARCA4缺失时其表达逆转的top基因,因为在这些条件下观察到不同的细胞活力表型(图4B和4C)。在PC9-OR和YU-005C细胞中,SMARCA4敲低引起的上调和下调基因在特定信号通路和细胞增殖和免疫信号相关的生物进程中富集(图4D)。此外,在PC9-OR细胞的耐药性相关DEG中,发现在SMARCA4敲低和奥西替尼处理后,20%的DEG被解除调节(图4E),其中包括分别参与细胞存活和异生代谢过程的基因,如BCL2或CYP26a1(图4E)。联合ATAC-seq,在PC9-OR和YU-005C细胞中,SMARCA4抑制和奥西替尼处理后,鉴定到了可及性和基因表达均降低的关键基因(图4F和4G)。一个子集属于AP-1 TFs家族(FOS和JUNB)以及参与癌症和增殖的其他基因,如HES1、EGR1和JAG1(图4G)。这些数据强调了SMARCA4缺失对维持抗性状态的影响,并确定了受其破坏影响的关键基因。
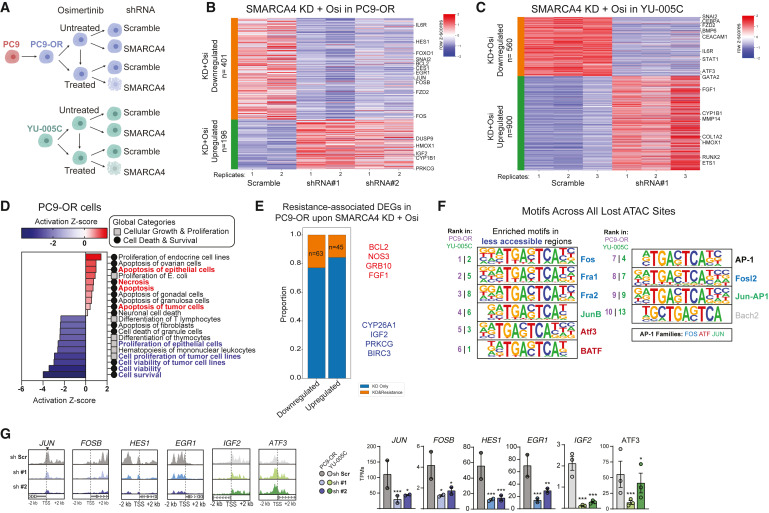
图4:奥西替尼耐药细胞系中SMARCA4抑制可调节耐药相关基因
5.药理学抑制mSWI/SNF ATP酶活性逆转EGFR突变癌细胞对奥西替尼的抗性
Cmp14是特异性变构SMARCA4/2 ATP酶的抑制剂。作者使用combenefit测定法测试几个药物协同作用(图5A)。作者先前的工作表明ERK再激活是EGFR-TKI治疗失败的主要决定因素,通过与MEK抑制剂联合治疗可以避免这种情况。在Cmp14存在或不存在的条件下,用奥西替尼和曲美替尼(OT)处理PC9*和PC9-OR*细胞,用于组合测定(图5A)。
combenefit分析揭示了OT和Cmp14之间的显著协同作用,其对PC9-OR*细胞具有特异性(图5A)。与单独OT处理相比,Cmp14额外添加增强了PC9-OR*中的细胞凋亡(图5B)。为确定在PC9-OR*细胞中观察到的药物协同作用背后的转录和染色质可及性变化,作者进行了RNA-seq和ATAC-seq实验(图5C)。对DMSO、OT和OT+Cmp14之间差异调节基因进行聚类分析,其结果显示四组关键基因(图5D)。C1包括DMSO组与Osi+Tram组中下调而在OT+Cmp14组中上调的基因, C2包含在OT处理激活但在OT+Cmp14时强烈下调的基因,C3包括其表达因OT处理而轻度降低,但因OT+Cmp14处理而强烈降低的基因, C4包括在对照和OT处理条件下都有活性,但与Cmp14联合后强烈下调的基因(图5D)。这些数据与ATAC-seq结合后揭示了与这四个聚类中基因相对应的基因座染色质可及性与基因表达变化相关(图5D)。接下来,作者对C1-C4转录反应进行表征,突出其“主要”靶标(具有与染色质可及性变化一致的靶标)和“次要”靶标(缺乏可及性一致变化的靶标)(图5E)。OT+Cmp14处理后下调的基因包括参与细胞周期和凋亡、细胞迁移和粘附、代谢过程和核受体通路因子,包括NRF2信号和毒素代谢,如EPHA4,RAC2,CDC25C和GPX2(图5D-5F)。上调基因(C1)包括参与蛋白激酶活性、免疫信号传导和核受体(如ATF3和CYP1B1)等过程基因(图5E和5F)。在8344个对OT+Cmp14表现出可及性降低的位点中,发现超过40%的位点在PC9-OR*细胞中含有BAF复合物峰(图5G,顶部)。此外,这些位点的一个子集(绿色圆圈)被定位到抗性状态下选择性上调但仅在OT+Cmp14联合处理时下调的基因(品红色圆圈)(图5G,底部)。这在CES1基因座上得到例证,在该基因座上,作者观察到在OR*(耐药)环境中BAF复合体的占有率和可及性增加,而在OT+Cmp14治疗后,这种情况显著降低(图5H)。CES1是一种关键的NRF2调节酶,它介导外源性代谢,并与肝细胞癌的化疗耐药性有关。
最后,作者旨在确定抵抗状态下only上调或下调的基因(即PC9-or*与PC9*)是否可以在OT+Cmp14联合治疗环境中相对于仅OT选择性逆转。在鉴定60个的基因中,其表达在抗性状态下下调,单独OT处理未能改变其表达,但OT+Cmp14联合处理(TGFA、GDF15和NDRG1)后其表达逆转(上调)(图5I)。同时,在76个基因中, OT+Cmp14联合处理(COL4A1、MAP2K6和EPHX1)中,其抗性相关表达被选择性逆转(下调)(图5I)。此外,作者还分析了一组基因,这些基因在PC9*细胞经OT处理后表达发生变化(上调或下调的基因),而在PC9-OR*细胞中也做出相应反应(图5J)。这些结果表明,抑制mSWI/SNF ATP酶与OT发生协同作用,部分通过重新联系染色质可及性来逆转耐药状态下的部分转录程序,使耐药PC9-OR*细胞对TKI处理重新敏感。

图5:靶向mSWI/SNF复合物ATP酶活性的药理学逆转EGFR突变癌细胞系亚群中的TKI耐药性程序
6.由mSWI/SNF复合物降低的活性氧与奥西替尼重新敏感有关
接下来,作者试图通过SMARCA4/2抑制或SMARCA4敲低来鉴定对奥西替尼重新致敏的EGFR突变细胞系潜在标志物或标志性特征。作者分析了对奥西替尼重新致敏的细胞系与未遵循SMARCA4 敲低或药物抑制细胞系之间耐药性DEG的差异。这揭示了PC9-OR(再敏化)细胞系与H1975-OR和HCC827-OR(未敏化)细胞株之间的差异,以及PC9-OR*(再敏化的)细胞系和HCC4006-OR(非敏化的)之间的差异(图6A和6B)。GSEA通路分析显示,相对于未致敏细胞系,再致敏细胞系PC9-OR和PC9-OR*中于PI3K信号传导和活性氧(ROS)通路等通路富集(图6B)。每个再致敏细胞系也表现出干扰素α/γ信号特异性阳性或阴性富集,表明细胞系特异性通路功能(图6B)。同时,为确定可能增强PC9-OR和PC9-OR*细胞对奥西替尼或OT处理再致敏反应的基因靶点,作者鉴定了PC9-OR*和PC9-OR细胞与HCC406-OR、H1975-OR或HCC827-OR相比 的DEG,其中特异性上调和下调的DEG(图6C)。某些DEG富集通路与MAPK信号传导和解毒有关,包括上调基因MAPK1、CRKL和CES1,以及下调基因PLK3和ARNT2(图6D)。
接下来,作者在PC9-OR*细胞、PC9-OR细胞和YU-005C细胞中研究与SMARCA4抑制或敲除后BAF介导的基因表达变化相关的染色质可及性变化基序(图6E和6F)。PC9-OR*细胞中与Cmp14协同的基因附近可及性降低的位点中,与AP-1因子和NRF2相对应的基序是参与协同反应的假定候选者(图6E),在PC9-OR和YU-005C细胞中SMARCA4敲低和奥西替尼处理后失去可及性位点的基序分析也显示NRF2是一个候选因子(图6F)。
NRF2信号通路负责在氧化应激条件下通过激活抗氧化反应清除活性氧。为验证SMARCA4/2介导的奥西替尼重新致敏、ROS和解毒通路之间的相关性,作者研究了奥西替尼如何影响PC9-OR和YU-005C TKI抗性细胞中的ROS水平。奥西替尼处理后,PC9细胞中ROS水平显著增加,这与NRF2信号整体减少一致(图6G、6H)。相反,PC9-OR细胞中基线ROS显著更高,奥西替尼没有深刻影响ROS水平,这与活性NRF2信号一致(图6G、6H)。在奥西替尼处理的PC9-OR细胞中敲低SMARCA4进一步增加ROS水平,同时降低NRF2通路活性,表明SMARCA4对于ROS中起着关键作用(图6H和6I)。与这些观察结果一致的是,SMARCA4敲除后,在奥西替尼治疗的肿瘤中,YU-005C皮下肿瘤中的ROS水平最高(图6J)。作者接下来研究NRF2在抗性表型中的功能。NRF2敲低降低了PC9-OR细胞对奥西替尼的敏感性,这证实NRF2是维持抗性的重要因子。此外,作者测试ROS存在细胞对奥西替尼反应中是否有直接影响。作者在EGFR突变肿瘤的组织微阵列中检测NRF2和SMARCA4的水平。这些肿瘤中SMARCA4水平与核NRF2呈正相关,进一步支持这两种蛋白质可能共同调节氧化应激(图6L)。这些结果证实了SMARCA4染色质重塑活性在通过NRF2激活控制奥西替尼诱导的耐药细胞氧化应激水平中的关键作用。
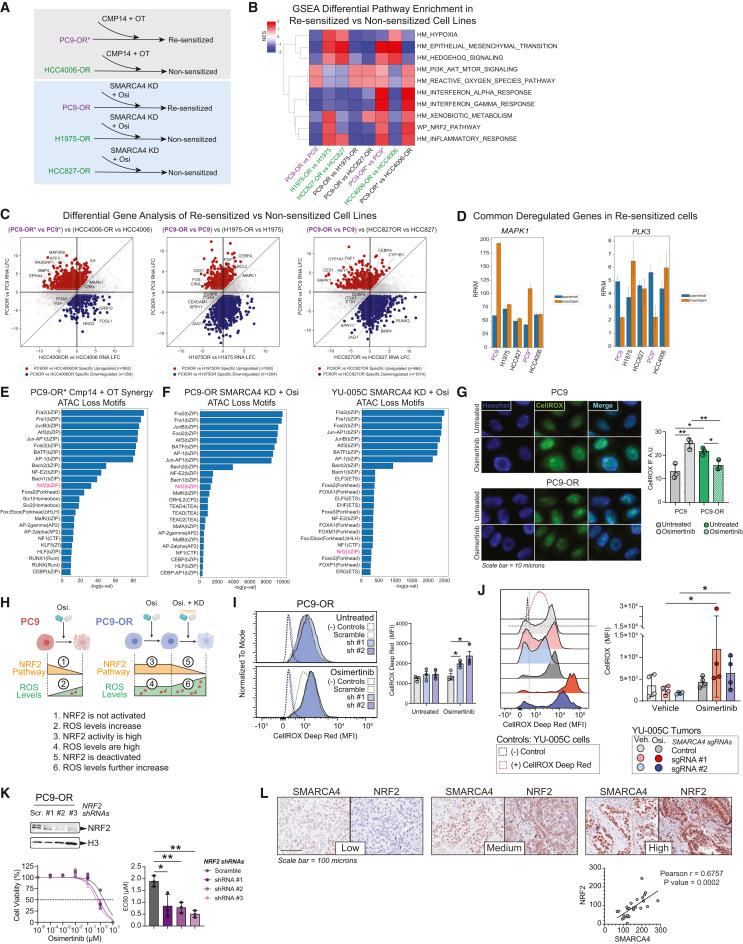
图6:奥西替尼抗性细胞系减敏揭示了mSWI/SNF复合物降低活性氧
7.奥西替尼抗性PDX小鼠模型中药理学抑制mSWI/SNF ATP酶活性降低肿瘤生长
接下来,作者探讨mSWI/SNF ATP酶活性的药理学抑制在SMARCA4依赖性YU-005患者衍生模型中的作用。YU005C细胞对单独的Cmp14或FHD-286(临床阶段双重SMARCA4/2=抑制剂)处理具有耐药性。然而,Cmp14和FHD-286均使YU005C细胞对奥西替尼敏感(图7A-7C)。因此,作者使用FHD-286在体内研究SMARCA4/2抑制与奥西替尼结合的潜力(图7D)。用载体、奥西替尼、FHD-286或奥西替尼和FHD-286组合处理荷瘤小鼠18天。虽然每种药物单独使用导致适度的肿瘤生长减弱,但奥西替尼+-FHD-286联合治疗显著抑制体内肿瘤生长(图7E)。
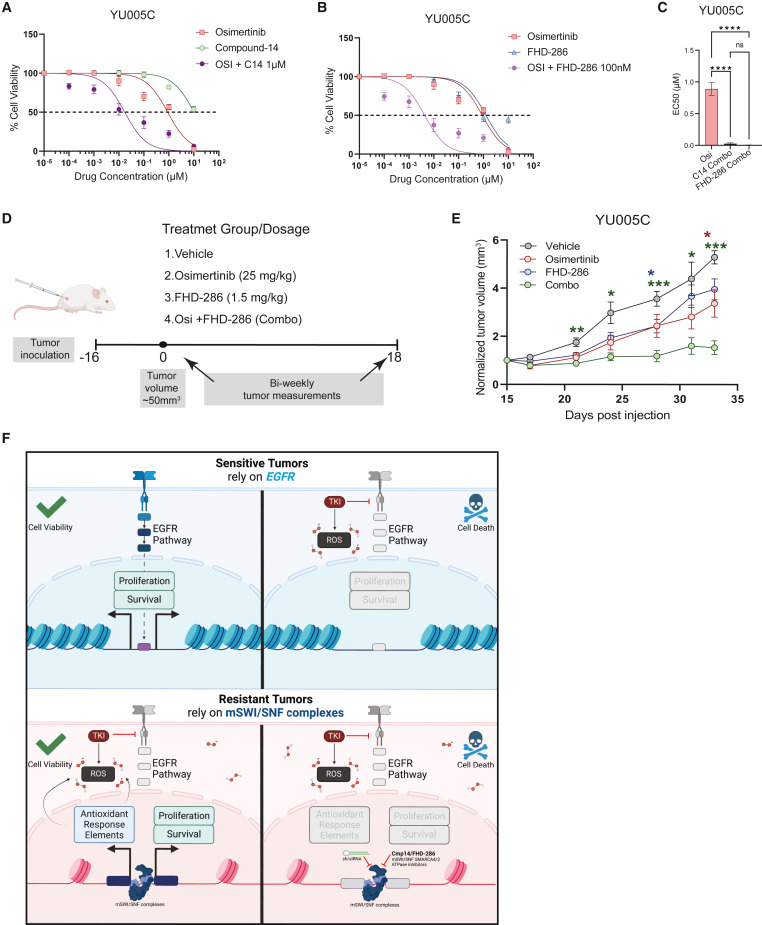
图7:mSWI/SNSF ATP酶活性的药理学抑制使患者来源的肿瘤对奥西替尼敏感
结论
基因敲低联合抑制剂抑制mSWI/SNSF ATP酶活性可使TKI耐药EGFR突变肺癌对奥西替尼重新敏感,并可增强细胞内活性氧水平且抑制肿瘤生长。
实验方法
CUT&RUN,ATAC-seq,RNA-seq,IHC,IF,细胞培养,药物实验,细胞凋亡检测,活性氧检测,细胞活力检测
参考文献
de Miguel FJ, Gentile C, Feng WW, Silva SJ, Sankar A, Exposito F, Cai WL, Melnick MA, Robles-Oteiza C, Hinkley MM, Tsai JA, Hartley AV, Wei J, Wurtz A, Li F, Toki MI, Rimm DL, Homer R, Wilen CB, Xiao AZ, Qi J, Yan Q, Nguyen DX, Jänne PA, Kadoch C, Politi KA. Mammalian SWI/SNF chromatin remodeling complexes promote tyrosine kinase inhibitor resistance in EGFR-mutant lung cancer. Cancer Cell. 2023 Aug 14;41(8):1516-1534.e9. doi: 10.1016/j.ccell.2023.07.005. Epub 2023 Aug 3. PMID: 37541244.