Cell热点:大脑皮层类器官联合多组学分析
要想充分发挥脑类器官研究人类发育的作用,就必须了解类器官是否精确复制了内源性细胞和分子事件,特别是因为类器官中细胞身份的获得可能会受到异常代谢状态的影响。本文展示了人类大脑皮层类器官发育的综合单细胞转录组、表观遗传组和空间图谱,包括超过61万个细胞,从神经祖细胞的生成到分化的神经元和胶质亚型的产生。本研究表明,无论新陈代谢状态如何,细胞多样化过程都与内源性细胞密切相关,因此,可以利用类器官图谱研究人类命运的决定。作者定义了器官发育过程中皮质细胞类型的纵向分子轨迹,确定了在细胞系建立过程中具有人类特异性作用的基因,并发现了人类胼胝体神经元的早期转录多样性。这些发现验证了人类类器官皮质发生综合图谱是研究人类皮质发育机制的首要资源。本文于2022年9月发表在《Cell》IF:64.5期刊上。
技术路线

主要实验结果
1、人类皮质类器官发育的单细胞水平转录组、表观基因组和空间图谱
为研究人脑类器官中皮质发生的分子特征和可重复性,构建了一个纵向单细胞图谱,包含类器官发育6个月期间的8个时间点,涵盖从早期祖细胞扩增到后期星形胶质细胞产生的过程(图1A),并在RNA、染色质和空间转录组学水平进行了分析。本文的转录组数据集包含本研究scRNA-seq检测出的532,414个细胞,并结合了作者之前发表的数据集,其来自多个时间点的218,240个新鉴定出的细胞,共计83个单独鉴定出的类器官,来自多个干细胞系和分化批次(图1A-1B)。表观基因组数据集包括38,017个核,包括单细胞ATAC-seq获得的转座酶可及染色质,并结合了作者之前发表的数据集,其包含体外培养1、3和6个月后类器官的11,551个新发现的细胞核,分别跨越了神经祖细胞的扩增、兴奋性神经元多样性的峰值以及星形胶质细胞和中间神经元的出现(图1C)。此外,纳入了通过SHARE-seq分析的42,810个细胞,SHARE-seq在从23日至3个月的4个时间点同时从同一单细胞采集了RNA-seq和ATAC-seq分析结果(图1D)。最后,空间转录组数据集包含10个类器官,这些类器官在体外1、2和3个月时通过Slide-seqV2分析得到(图2),涵盖兴奋性神经元的出现和扩张,这些神经元在体外3个月时类器官的多样性最高。这些数据提供了人类皮质类器官发育的综合多组学分子图谱.
作者评估了这些细胞产生的纵向事件在所有类器官的发育过程中可重复性,通过计算每个采样时间点的调整后互信息(AMI)得分,衡量了存在的细胞类型比例与单个起源的类器官之间的依赖关系(图1E)。在各年龄段和细胞系中,大多数重复的 AMI 分数都与两个已发表的内源性人类皮层数据集和一个新生成的胎儿数据集相当。并且总体而言,scRNA-seq和scATAC-seq定义的细胞类型之间存在良好的一致性,AMI评分表明,个体类器官之间的表观遗传变异性与转录变异性处于相同的低水平(图1F)。表明这些随着时间延伸的类器官数据也在多组学层面展现出了细胞分化的可重复性。
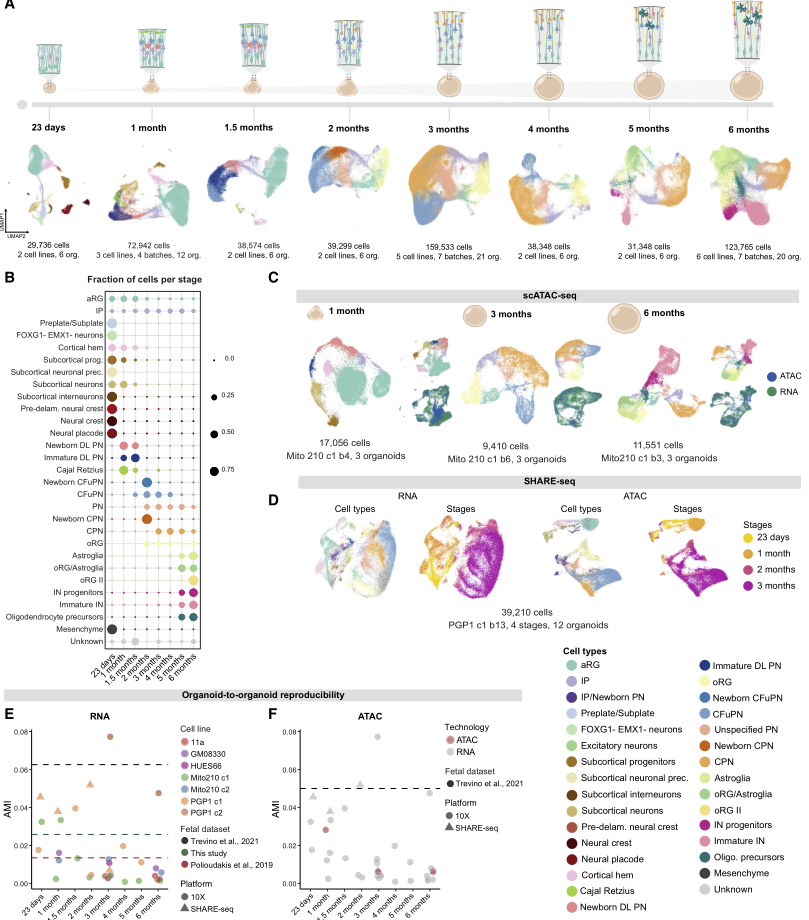
图1 皮质类器官发育的单细胞转录组学和表观遗传学景观
2、类器官发育过程中细胞类型的空间组织
为在其原始空间背景下分析分子和细胞事件,在1个月、2个月和3个月时使用Slide-seqV2进行空间转录组分析(图2)。1个月时,顶端放射状神经胶质(aRG)祖细胞簇占据类器官的中央和周边区域(图2A-2E),Cajal Retzius细胞、中间祖细胞(IP)、新生深层(DL)投射神经元(PN)和未成熟的DLPN相对于aRG位于表层。有趣的是,皮质边缘区和皮质下区的细胞在很大程度上没有与皮质细胞类型混合。2个月时,aRG在主要由“非特指PN”群体组成的核心周围形成了一圈玫瑰花结样结构(图2F-2H)。其余的皮质细胞类型主要位于aRG的浅表。外放射状胶质细胞(oRG)相对于aRG进行了表面定位(图2H),反映了这些细胞在内源性组织中的相对位置。到3个月时,可识别的结构如玫瑰花结丢失(图2I-2K),与文献中研究结果一致。类器官的内核主要由“非特指PN”和aRG构成(图2K)。这些数据显示了皮质类器官中细胞类型的动态变化和优先定位,反映了体内观察到的情况。
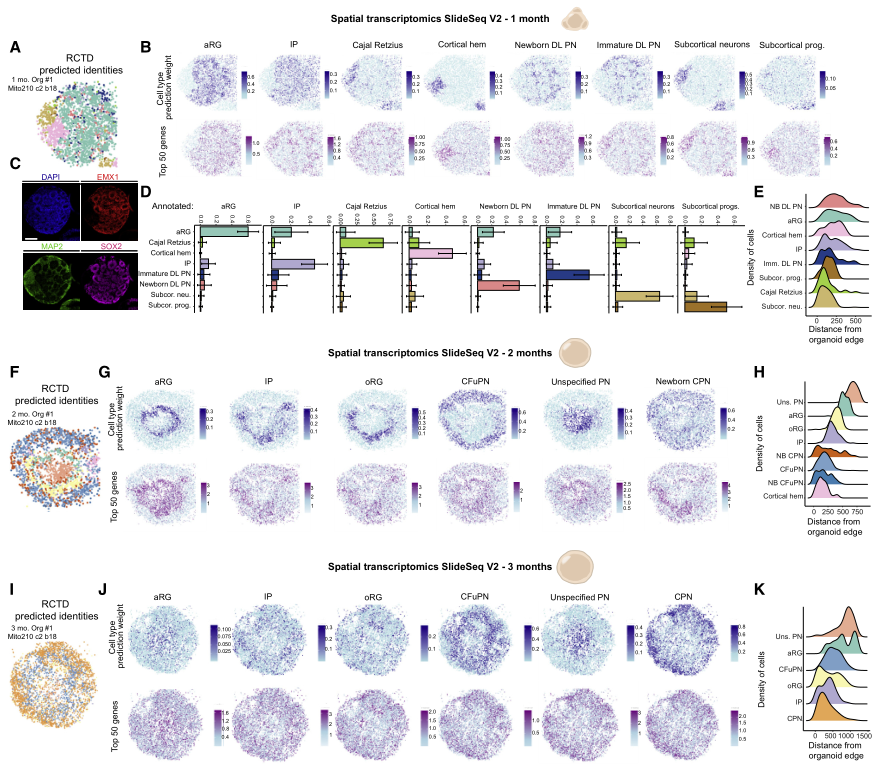
图2 发育的皮质类器官的空间转录组景观
3、皮质类器官捕获具有细胞类型和时间特异性的细胞身份获得的纵向胎儿程序
先前的研究表明皮质器官组织和内源性人类胎儿组织有相似之处,这些相似性是否扩展到所有细胞类型和发育的所有步骤还有待探索。为解决这一问题,作者首先确定了每个阶段所有皮质细胞类型的基因表达标签(图3A-3B)。接下来,通过在每种细胞类型的可及染色质区域鉴定转录因子(TF)基序富集,并将其与同源TF的表达进行比较,研究了假定的细胞类型特异性调节机制。结果证实了已知的发育阶段和细胞类型合适的TF及其基序是与相关的细胞群相关(图3C)。
接下来,试图确定类器官中识别皮质细胞类型的分子信号与内源性胎儿皮质中定义皮质细胞类型的分子信号有多密切。将类器官数据与两个已发表的scRNA-seq数据集中的91,844个图谱和60806个单核RNA测序集进行了整合(图3D)。以两种方式评估相似性,这两种方式都表明类器官和胎儿皮质细胞类型之间高度一致。
首先,应用了基于随机森林分类器的方法。每个阶段的类器官细胞主要由在胎儿细胞上训练的分类器分配到相应的内源性细胞类型,反之亦然(图3E-3F)。在用类器官细胞训练的分类器中,很少有胎儿细胞被分配给aRG和“未指定的PN”类器官细胞(图3F),这表明这两个群体与内源性细胞类型的关系不太密切。
其次,使用秩-秩超几何重叠的方法对类器官和胎儿细胞中每种皮质细胞类型分别定义的分子特征进行了两两比较(图3G)。该方法还显示了类器官和相应内源性胎儿细胞类型之间的高一致性表达特征(图3H-3K)。
重要的是,作者发现类器官“未指定PN”的分子标签,虽然与Polioudakis等人(2019)的胎儿迁移ExNs、Trevino等人(2021)的GluN 1和5簇以及本文胎儿数据集的CPN最接近,但总体上显示出相对较弱的一致性,主要是由共同的下调基因驱动(图3J)。类似地,类器官aRG的标记显示,从体外2个月开始的任何时间点,上调基因与任何胎儿标记都没有大量重叠(图3K)。因此,尽管这两种细胞类型显示出与内源性细胞的一些总体转录相似(图3E),但它们的转录特征与这一胎龄范围内细胞类型特异性胎儿特征的匹配性相对较弱。
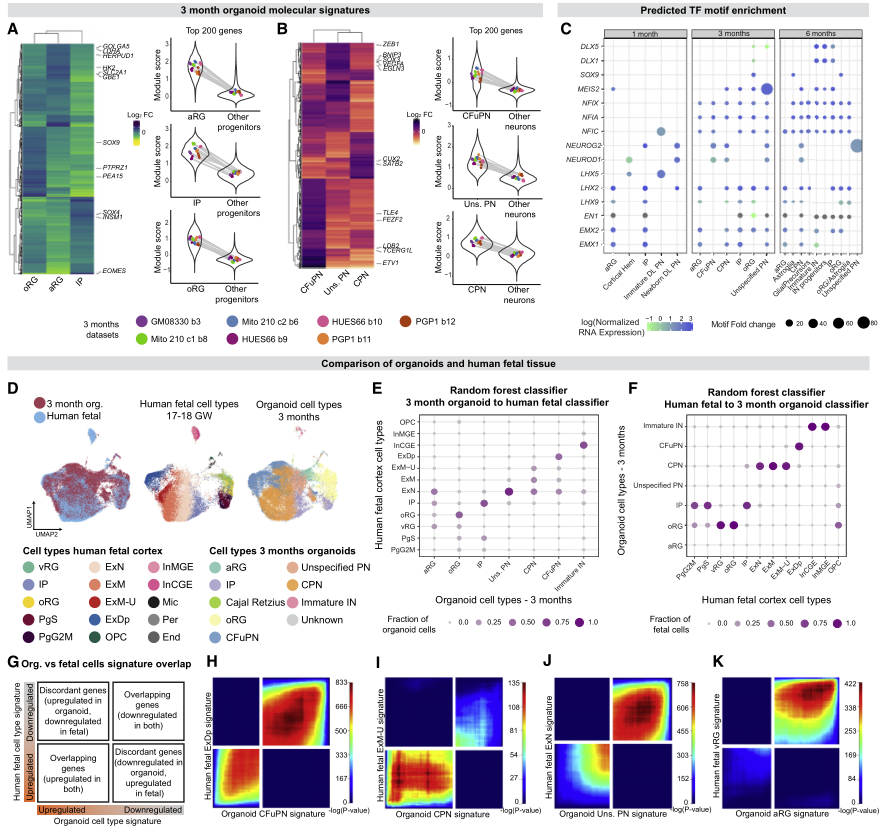
图3 在具有细胞类型特异性的皮质类器官中建立细胞身份获取的纵向胎儿程序
4、人类皮质类器官细胞的类型在很大程度上不受代谢状态的影响
WGCNA确定了一个模块,其中包括从体外1.5个月开始的糖酵解相关基因,从体外2个月开始,该模块在与胎儿细胞最不相似的两个细胞群中特异性富集,即aRG和“未指定PN”(图4A和4B)。在类器官发育的多个阶段,“未指定的PN”和aRG以及其他类型的祖细胞都富集了糖酵解和缺氧基因集(图4C和4D)。然而,这与在内源性人类胎儿祖细胞子集中观察到的这些基因集的富集是相似的(图4E)。因此,在正常皮质祖细胞发育过程中观察到的代谢状态也在类器官中相应的细胞类型中观察到。
对器官组织与内源性胎儿细胞在代谢基因表达方面的差异程度进行的更广泛研究显示,所有细胞类型之间的差异都很有限。作者采用三种互补方法: Compass,该方法利用通量平衡分析从 scRNA-seq 数据中模拟单细胞的代谢状态;RRHO2,识别富集在类器官中上调而在胎儿细胞中下调的基因的过程;以及 MSigDB 代谢基因组的差异表达。在所有三项分析中,类器官和胎儿细胞显示出相似的结果,只有少数代谢通路,特别是糖酵解和氧化磷酸化,在类器官中富集(图4F-4G)。值得注意的是,Compass显示类器官和内源性胎儿细胞很好地聚集在一起,不能根据它们的代谢通量进行区分(图4F-4G)。
代谢基因在aRG和“非特指PN”群体中的富集可能会干扰类器官细胞类型与其内源性对应的排列,正如文献里其他皮质类器官模型所建议的那样。因此,作者从用于训练随机森林细胞类型分类器的可变基因中系统地删除了38个MSigDB代谢基因中的每一个,并评估了对结果细胞分类的影响。对于绝大多数基因集,去除这些基因并没有显著改变分配给每种细胞类型的细胞数量(图4H-4J)。只有两类代谢过程影响分类:在用3个月的类器官细胞训练的分类器中,去除与葡萄糖代谢和缺氧相关的基因集显著增加了分配到“未指定PN”和aRG细胞类型的胎儿细胞数量(图4H-4J)。没有其他细胞类型显示分配增加。重要的是,与凋亡和氧化磷酸化等其他基本亚细胞过程相关的基因未显著改变任何胎儿细胞类型向类器官细胞类型的分配(图4H)。

图4 人类皮质类器官的细胞类型在很大程度上不受代谢状态的影响
然后,作者利用空间转录组学发育图谱来研究皮质类器官细胞的地形位置和代谢状态之间是否存在关联。2个月和3个月的类器官内部区域主要由aRG和非特指PN组成(图2F-2K,图5A),这两个群体的代谢状态影响了细胞身份的分配。值得注意的是,aRG在1个月和2个月之间变得更加集中,这与我们的发现一致,即aRG的身份在1.5个月开始受到影响(图5B)。此外,虽然38个MSigDB代谢通路基因集的表达在类器官的直径中是恒定的,但与缺氧和糖酵解相关的通路在类器官中心的细胞中富集(图5C-5D)。值得注意的是,位于类器官内部区域的细胞显示出更高的糖酵解和缺氧基因的表达,这与细胞的特性无关(图5E)。总之,分析表明,在目前的类器官模型中产生的绝大多数皮质细胞类型的鉴定不受不同的代谢状态的影响,这只影响了两种细胞类型(aRG和“非特指PN”)的鉴定,在2-6个月时仅占类器官细胞的3% - 15%。
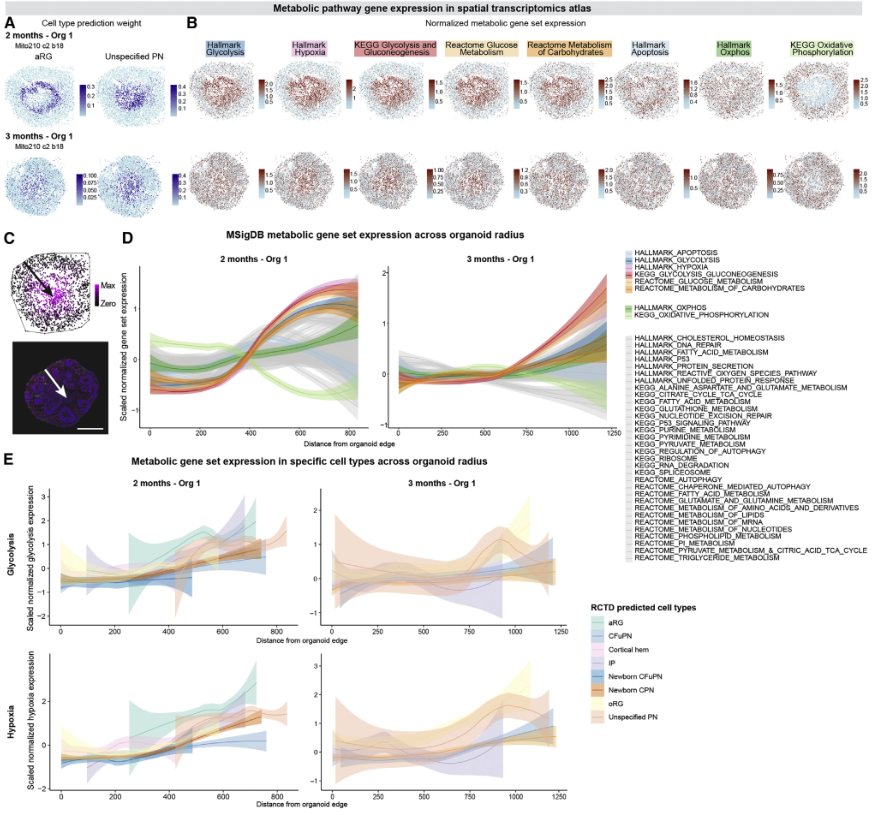
图5 代谢受损的细胞存在于人类皮质类器官的一个有限的中心区域
5、人类个体皮质细胞类型发育的分子逻辑
前文的结果表明,可以在类器官中建立适当的细胞多样性,并且细胞身份在很大程度上不受代谢状态的影响。因此,作者试图应用这种类器官发育的分子图谱来理解人类皮质中细胞谱系的建立,以及与谱系决定相关的转录事件。作者利用459,711个皮层细胞(不包括未指定的PN)推断出发展轨迹。为了突出不同时间细胞类型之间的关系,通过转录相似性连接了来自相同和连续时间点的细胞簇,同时保留了细胞的已知时间阶段(图6A)。有趣的是,在较晚的时间点,oRG同时连接到神经元和胶质祖细胞(图6A)。作为一种补充方法,作者应用轨迹推断算法URD来生成基于伪时间有序细胞转录相似性的分支轨迹树(图6B)。将根定义为存在于体外23天的最早阶段的aRG,而尖端定义为每个皮质谱系的终末细胞类型。在树中建立的顺序反映了细胞的阶段和分化状态,基础祖细胞在aRG之后,然后分化为神经元(CFuPN,CPN)和胶质(星形胶质细胞,少突胶质细胞前体细胞)谱系。
使用该树绘制了发育过程中的动态表达变化(图6C-6D),并确定了与人类皮质神经元亚型相关的谱系特异性基因。为此,将这一人类皮质树与已发表的小鼠发育皮质URD树进行了比较(图6E)。这一分析揭示了人类类器官树中的谱系特异性基因,而这些基因在小鼠树中表现出不同的表达模式(图6F)。例如,SORCS1在人类类器官树中与CFuPN谱系相关,但在小鼠的多个皮质投射神经元中表达。同样,PIK3R1在人类中与CPN谱系相关,但在小鼠中表达更广泛。
为提取与人类细胞命运决定相关的分子程序,确定了与人类类器官URD树中每个谱系分叉相关的top基因和TF(图6D)。为了区分控制这些分叉的哺乳动物和人类特有的共享程序,比较了小鼠中与同源分叉相关的TF。值得注意的是,一些被预测在人类神经元命运中发挥作用的候选人类调节因子没有1:1的小鼠同源物,包括ZNF26和ZNF37A(图6G)。

图6 人类皮层细胞类型的分子发育
6、人类胼胝体投射神经元多样性在胚胎发育过程中出现
作者检查了树的终末神经元细胞类型,以研究在成年期CPN基因表达的异质性是否在开发的早期阶段就已经可以检测到。5种不同CPN类型的标记基因在胎儿和类器官CPN的发育早期都有表达,CPN中高表达的标志物也在类器官中高表达,反之亦然(图7A)。为评估成年期发现的分子多样性是否也可在早期CPN中观察到,在成年CPN中确定了5个共变基因程序(模块),大致代表每种成年CPN类型(图7B和7C),并在胎儿和类器官CPN中对它们进行了评分。这5个模块在胎儿和类器官CPN中均有表达。虽然它们的表达并没有像在成年期中那样将发育中的CPN细分为5个亚群,但它们开始将早期发育的CPN细分为5个亚群,其中模块5表现出最独特的模式(图7D和7E)。因此,虽然成人CPN的完整表达多样性还没有出现,但数据表明CPN的分化是一个开始于早期发展阶段的过程。
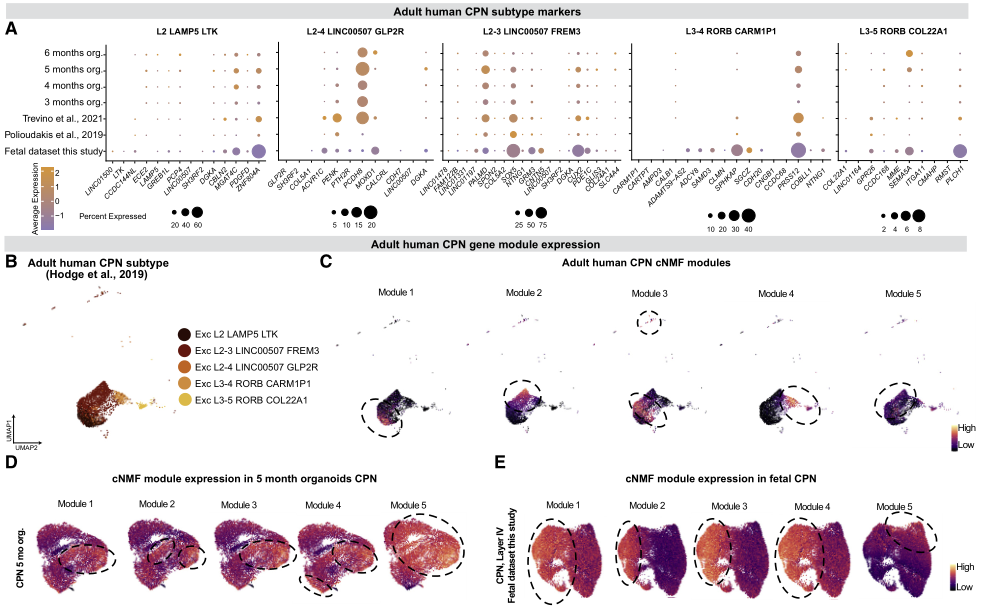
图7
实验方法
多能干细胞培养,类器官分化,人胚胎脑组织分离,免疫组织化学,低氧探针,免疫荧光,脑类器官分离,scRNA-seq及其生物信息学分析,单细胞ATAC-seq及其生物信息学分析,SHARE-seq和bulk ATAC-seq及其生物信息学分析,Slide-seqV2及其生物信息学分析,单核RNA测序及其生物信息学分析,大脑皮层类器官和人胚胎的细胞类型比较和匹配分析,WGCNA分析,代谢活动评价,皮质类器官和全脑类器官的比较,代谢基因对细胞分类的影响分析,与代谢相关的表观遗传变化分析,轨迹分析,人类胼胝体投射神经元多样性分析,量化和统计分析
参考文献
Uzquiano A, Kedaigle AJ, Pigoni M, Paulsen B, Adiconis X, Kim K, Faits T, Nagaraja S, Antón-Bolaños N, Gerhardinger C, Tucewicz A, Murray E, Jin X, Buenrostro J, Chen F, Velasco S, Regev A, Levin JZ, Arlotta P. Proper acquisition of cell class identity in organoids allows definition of fate specification programs of the human cerebral cortex. Cell. 2022 Sep 29;185(20):3770-3788.e27. doi: 10.1016/j.cell.2022.09.010. PMID: 36179669; PMCID: PMC9990683.