孕激素受体通过RUNX转录因子相互作用和染色质重塑介导排卵转录
孕激素受体(PGR)在生殖组织中扮演多种角色,从而调控哺乳动物的生育能力。在卵巢中,通过对一组独特基因的转录调控,迅速、急性诱导PGR是排卵的关键决定因素, 最终导致卵泡破裂。然而,关于PGR在排卵中的专门功能的分子机制尚不清楚。我们通过组合ATAC-seq、RNA-seq和ChIP-seq分析,在野生型和特定异构体PGR敲除小鼠中,建立了详细的PGR作用基因组谱系。我们证明,刺激排卵迅速重塑了三分之二的染色质可及性,与基因表达的改变相关。我们观察到,卵巢特异性的PGR作用与RUNX转录因子的相互作用有关,70%的PGR结合区域也与RUNX1结合。这些转录复合物将PGR结合到近端启动子区域。此外,PGR与典型的NR3C基序的直接结合使染色质具有可及性。所有这些PGR作用共同介导了必需的排卵基因的诱导。我们的发现突出了一种新的针对排卵的PGR转录机制,为不孕不育治疗或阻止排卵的新避孕药提供了新的靶点。
图形摘要

该研究于2023年4月发表发表在《Nucleic Acids Research》,IF:14.9。
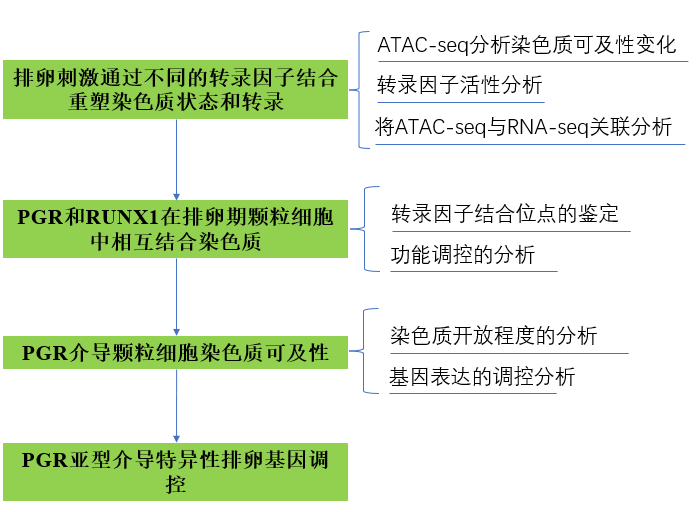
技术路线
结果
1、排卵刺激通过不同的转录因子结合重塑染色质状态和转录
通过hCG注射小鼠诱导排卵提供了一个体内模型,用于研究颗粒细胞对排卵信号的快速反应。比较ATAC-seq揭示了染色质可及性和排卵转录因子结合的动态变化。严格的ATAC-seq峰值调用标准确定了总共71,287个可访问的染色质峰,其中三分之二的峰在黄体生成素(LH)刺激后的6小时内发生了改变,有28,729个位点(40.3%)受LH诱导,21,257个位点(29.8%)显著抑制,21,301个位点(29.8%)可及性无变化(FDR ≤ 0.05,图1A)。LH介导的可及性变化不仅发生在启动子区域,还发生在远端增强子和基因区域(图1B)。
为了预测参与LH介导的开放染色质位点可及性变化的关键转录因子的活性,进行了转录因子结合模体的差异分析(图1C)。许多已知的排卵转录因子(如JUN/FOS、RUNX、CREB和CEBP)的模体在LH刺激后开放染色质峰中的结合分数显著增加,而CTCF、NR5A和ESRR的模体则富集于LH前开放染色质位点。有趣的是,NR3C家族的典型模体,包括孕激素受体(PGR)、糖皮质激素受体(NR3C1/GR)、醛固酮受体(NR3C2/MR)和雄激素受体(NR3C4/AR),在LH刺激前的开放染色质中显示出显著较高的结合分数。为支持这些发现,使用HOMER进行的新生模体富集分析也在LH诱导的开放染色质区域中鉴定出了JUN/FOS、RUNT和CEBP的模体,但没有在LH抑制的开放染色质区域中发现(图1D)。NR5A和GATA家族的模体在所有ATAC-seq峰集中同样富集。重要的是,在LH抑制的子集中仅发现与NR3C成员结合的模体最匹配,与差异转录因子结合分析模式一致。转录因子直接结合到这些模体的证据可在聚合的足迹图中观察到(图1E)。显示与转录因子结合到这些位点相关的足迹模式,可以观察到中度至深度的凹陷(JUN/FOS、CTCF、NR5A)和较浅的模式(RUNT、NR3C),暗示这些转录因子停留时间的差异。JUN/FOS足迹仅在LH刺激后才明显,而NR3C足迹的证据在未刺激的细胞中最明显。
通过对体内LH刺激前或LH刺激后8小时收集的颗粒细胞进行RNA-seq分析,将染色质可及性谱系与基因表达变化相关联。共鉴定了2088个差异表达基因(DEG),其中52%受LH刺激上调(图2A)。其中包括许多已知与排卵相关的基因,如Ptgs2、Pgr、Runx1、Runx2和Adamts1。使用Ingenuity Pathway Analysis(IPA)软件预测LH DEG的潜在上游调控因子,确定了一系列转录因子,其中许多也是对LH激增作出反应的DEG,包括PGR(log FC = 6.4)、RUNX1(log FC = 4.6)、CEBPβ(log FC = 2.5)和JUNB(log FC = 2.1)。值得注意的是,除了PGR外,这些转录因子的结合模体也显示出LH刺激后的可及性增加(图1C、D)。通过比较LH DEG和这些基因启动子(TSS上游3 kb内)的LH依赖性染色质可及性,展示了排卵前颗粒细胞中启动子可及性与下游基因表达之间的全局关系。69.3%的LH驱动DEG(2088个基因中的1447个)在启动子可及性发生显著变化时显示出转录变化(图2B,红色符号)。LH诱导的基因的启动子可及性大多增加,而LH抑制的基因的可及性较低(Spearman相关系数=0.4297)。作为示例,Cxcr4和Inhbb是两个分别上调和下调的基因,其表达模式与基因启动子处的ATAC-seq信号明显相关(图2C)。然而,在某些情况下,差异调节的基因具有一直可及的启动子,如Cited1。LH诱导的启动子可及性高的转录活跃基因主要与排卵相关的生物过程相关,包括血管生成、MAPK信号通路和炎症应答。总之,这些结果表明,作为对LH激增的全局染色质重塑,很可能通过激活一组特定的转录因子,驱动与LH触发的排卵相关的基因表达谱系。
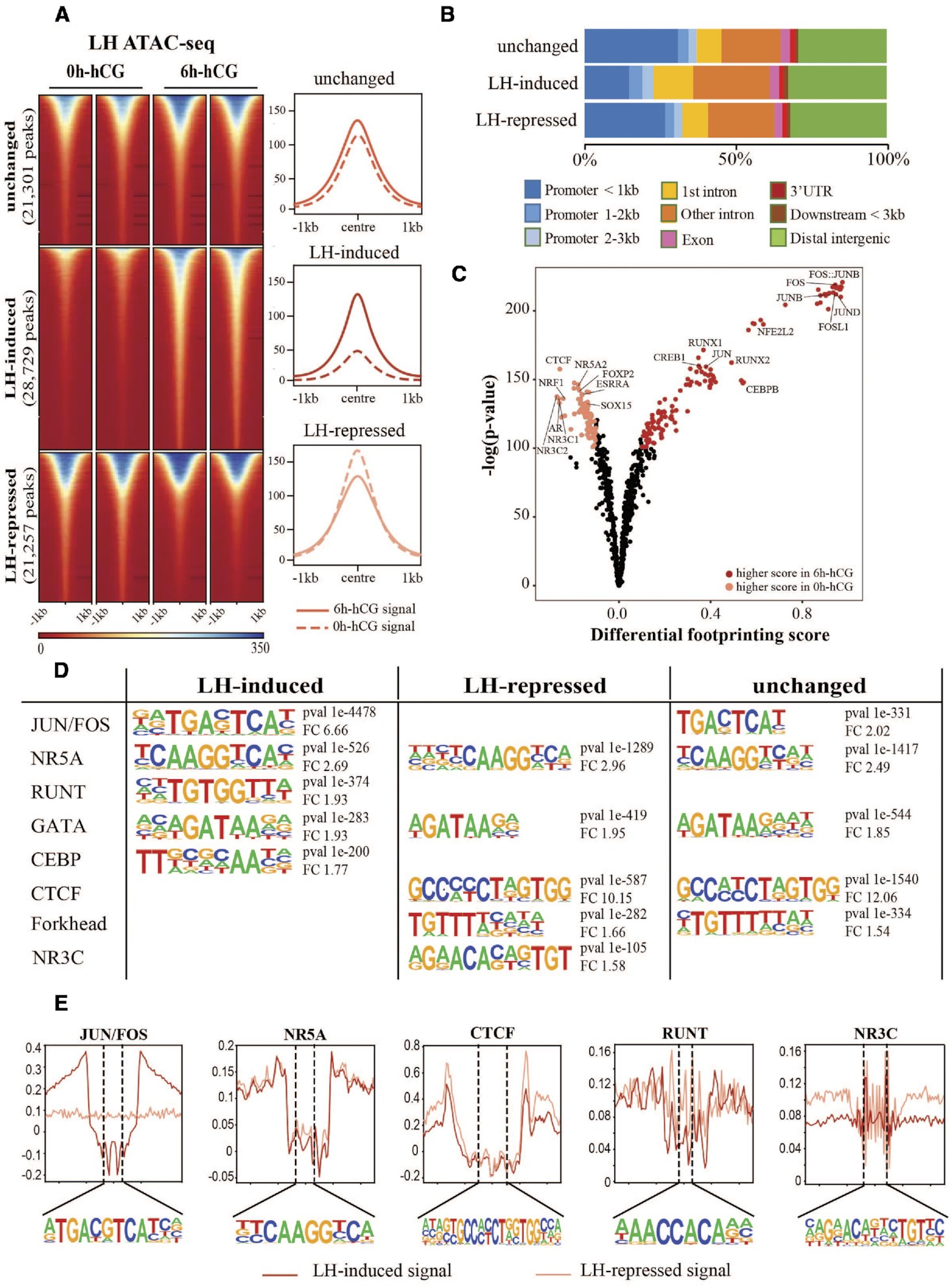
图1.人绒毛膜促性腺激素治疗后颗粒细胞染色质景观的整体变化
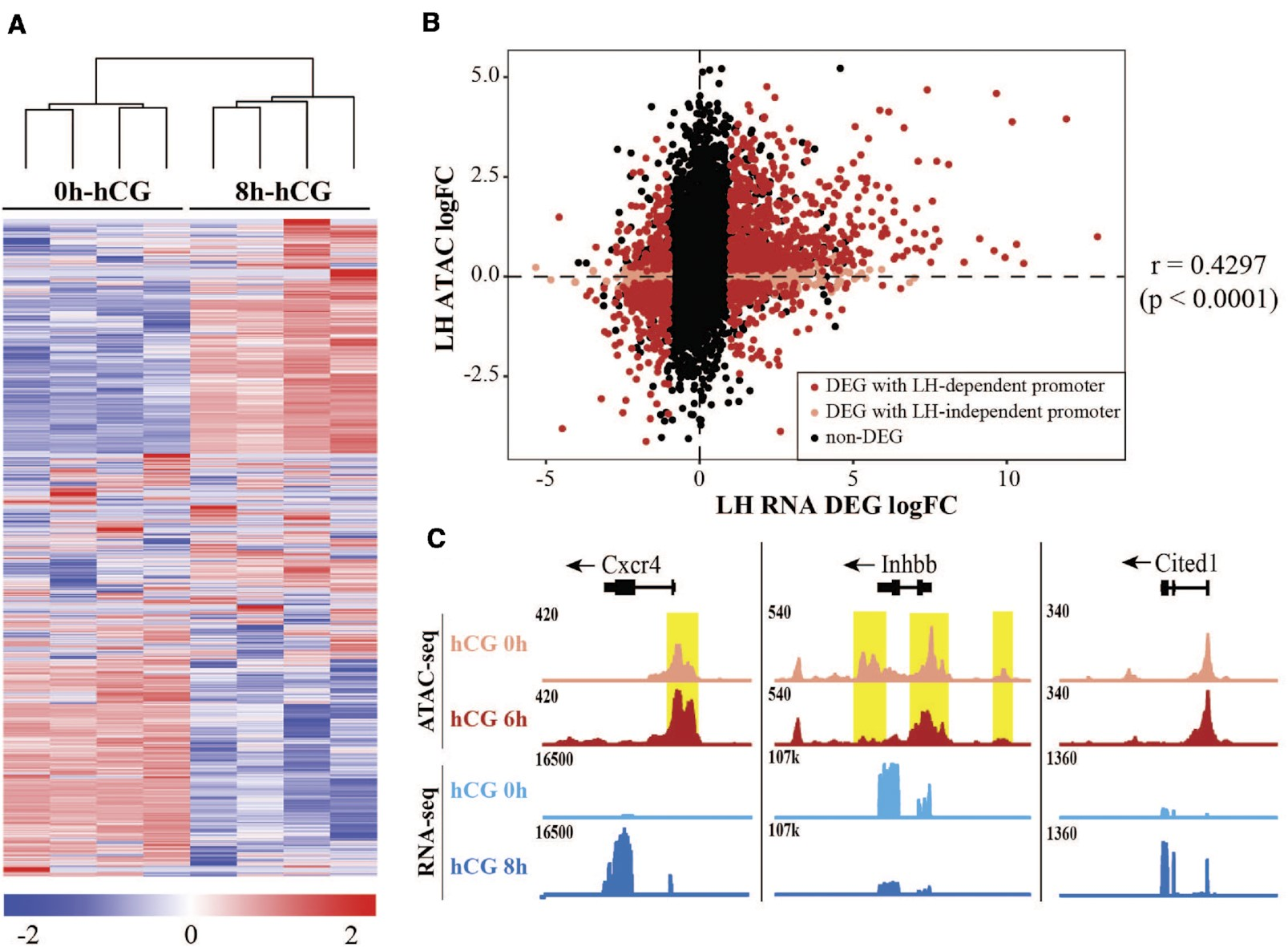
图2.hCG治疗后颗粒细胞的整体转录变化。
2、PGR和RUNX1在排卵期颗粒细胞中相互结合染色质
RUNX1在排卵中具有功能重要性,并且RUNT基序在围绝经期颗粒细胞的PGR ChIP-seq峰值中高度富集。此外,我们发现在LH刺激后获得染色质可及性的区域含有富集的RUNT基序(图1C)。RUNX1和RUNX2的表达在颗粒细胞中对LH激素的刺激作出反应时被诱导,并且在颗粒细胞特异性Sf1-cre RUNX1 KO小鼠中排卵被延迟。为了确定RUNX1是否是介导排卵基因表达的转录调控机制的关键因素,我们使用ChIP-seq在体内hCG处理前或处理后的6小时内定义了颗粒细胞中RUNX1的全基因组靶点。在LH激素释放前就检测到全局RUNX1-染色质结合,但在LH激素释放后也被7倍诱导(16065个LH诱导的峰值)(图3A)。LH激活的RUNX1结合与LH诱导的开放染色质强烈相关(通过ATAC-seq定义)。通过比较RUNX1和PGR的ChIP-seq,我们在对LH刺激做出反应的颗粒细胞中确定了RUNX1和PGR之间的明显关系。PGR和RUNX1在100 bp内共享了大量结合位点,其中9704个染色质位点(总PGR的70%或总RUNX1结合位点的52%)同时结合了PGR和RUNX1(图3B)。在这些共享的PGR/RUNX1结合位点中,有9288个位于同一时间点鉴定的ATAC-seq开放染色质位点中。将转录活跃的RUNX1和PGR cistrome分为每个转录因子共享或only结合的部分,发现没有RUNX1共结合的PGR结合位点与基因启动子的关联程度相对较低。在PGR/RUNX1共结合位点中,靠近启动子的结合(在TSS的1 kb范围内)是PGR的主要特点(图3C)。PGR和RUNX1结合位点的显著高度重叠,再加上我们先前报告的颗粒细胞PGR ChIP-seq中富集的RUNX1结合基序,暗示了PGR以先前报道的颗粒细胞特异性能力通过与RUNX1共结合来靶向近端启动子。这种依赖关系不是相互的,因为RUNX1显示出独立于PGR共结合的近端启动子占据的优势。为了测试LH诱导的RUNX1染色质结合是否依赖于PGR,我们比较了在PGR重叠位点与独立于PGR的位点上LH诱导的RUNX1结合强度。在RUNX1富集水平上没有观察到差异,这表明PGR不太可能在这些共享位点上发挥RUNX1的连接或招募作用。
通过将具有PGR和/或RUNX1结合的基因与我们的LH响应性差异表达基因列表进行比较,展示了PGR和RUNX1在基因表达中对排卵信号的重要性(图3D)。值得注意的是,所有LH介导的差异表达基因中,至少62.4%的基因在其近端启动子(在3 kb范围内)上具有PGR或RUNX1结合,与LH抑制的差异表达基因相比,LH诱导的差异表达基因更频繁地被PGR和/或RUNX1结合。其中,大多数(55%)RUNX1结合的LH差异表达基因和大多数(87%)PGR结合的LH差异表达基因实际上都被两个转录因子共结合,这意味着PGR和RUNX1在目标启动子上的同时相互作用是它们各自在基因调控中的主要机制。在排卵期颗粒细胞中的许多已知的PGR和RUNX1靶基因的启动子中可以看到PGR/RUNX1的相互染色质占据(Rgcc,Adamts1和Cstl)(图3E)。PGR/RUNX1的合作还可以通过形成与调控基因的近端启动子接触的远端增强子来调控靶基因的表达。为了研究这一点,我们整合了公开可用的小鼠颗粒细胞Hi-C的染色质-染色质相互作用数据。Hi-C确定了与516/2088个LH差异表达基因的TSS相关的远端染色质相互作用,其中至少28%被PGR和/或RUNX1结合(图3D)。与PGR和RUNX1在近端启动子上的结合不同,PGR/RUNX1在远端增强子上的结合与LH诱导和LH抑制的基因表达同样相关。以PGR靶基因Adamts1为例,它显示了强烈的PGR/RUNX1共结合,位于通过小鼠颗粒细胞中的Hi-C以及人类卵巢中的启动子捕获-C指示的远端区域与Adamts1 TSS相互作用(图3F)。
基序富集分析显示,期望的典型RUNT基序在RUNX1基因组中高度且显著富集(相对于背景的3.5倍,P值为1e-787),以及PGR基因组中(相对于背景的2.8倍,P值为1e-370)(图3G)。NR3C基序在RUNX1结合的基因组中也富集(相对于背景的2倍,P值为1e-25)。这种富集仅限于PGR/RUNX1共结合位点。其他富集的非典型基序包括与JUN/FOS、CEBP和GATA转录因子对应的基序。
通过近距离连接测定(PLA)证实了PGR和RUNX1在小鼠颗粒细胞核中的密切物理相互作用,这些细胞在模拟体内排卵刺激的情况下经过hCG和R5020处理长达8小时(图4A)。PLA信号显示LH刺激迅速短暂诱导了PGR/RUNX1复合物,表明在刺激前大部分时间不存在的PGR/RUNX1相互作用在刺激后4小时内增加。此外,通过免疫沉淀结合质谱法在PGR表达的人类颗粒细胞中,根据对R5020处理的响应,也表明了RUNX1和PGR之间的物理相互作用(图4B)。PGR还可以与其他RUNX成员相互作用,正如在暴露于排卵刺激的颗粒细胞中的PGR/RUNX2 PLA所示(图4C)。

图3.在排卵周围颗粒细胞中,PGR和RUNX1共有共同的染色质靶点。
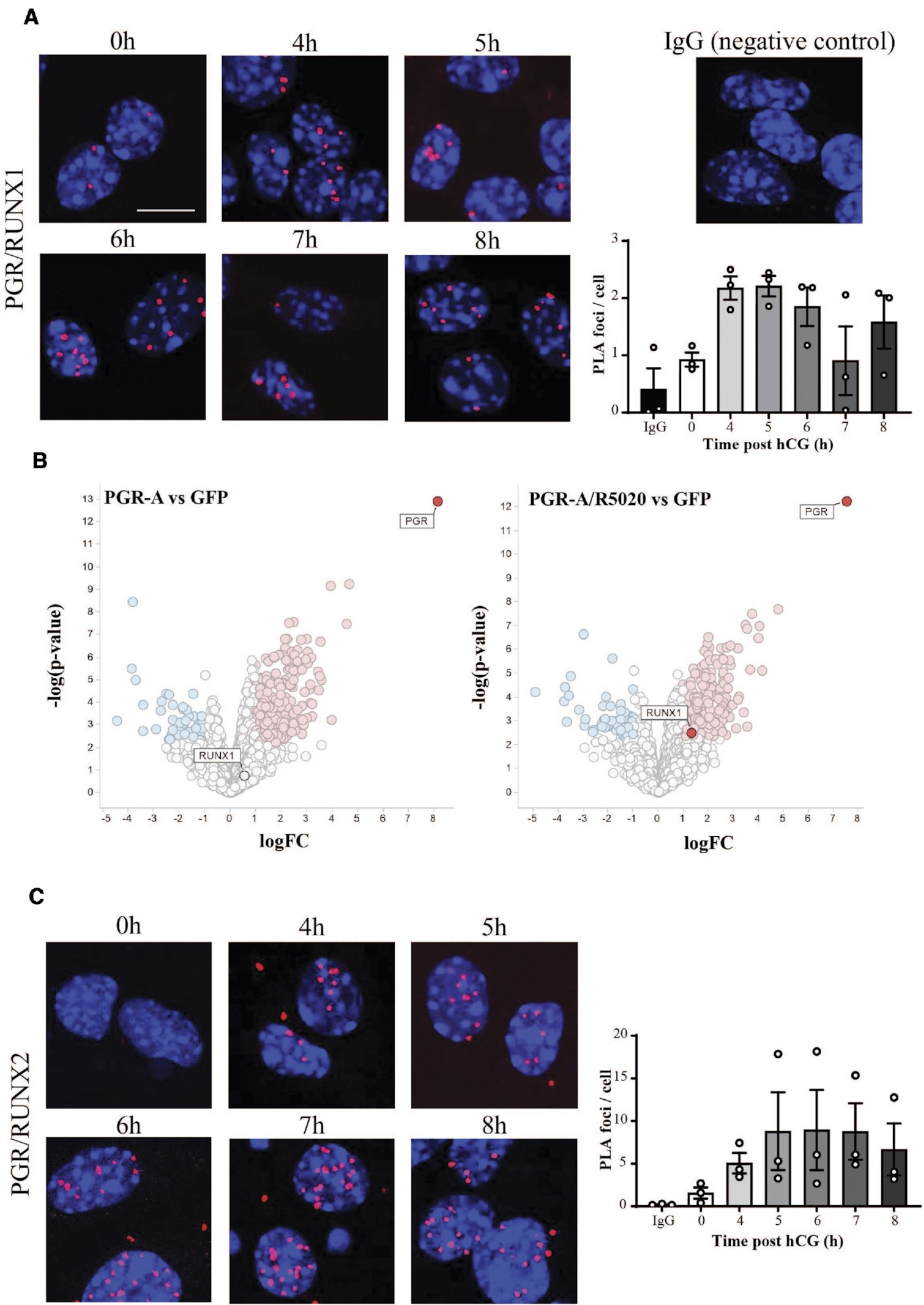
图4.颗粒细胞中的PGR与RUNX转录因子相互作用,以响应黄体生成素激增。
3、PGR介导颗粒细胞染色质可及性
PGR结合区域主要与转录活性染色质重叠,如PGR和H3K27ac ChIP-seq峰值重叠75%,以及90%的PGR结合位点与ATAC-seq峰值重叠。此外,在激素刺激的颗粒细胞中还展示了PGR与组蛋白乙酰转移酶CBP/p300之间的相互作用。通过对PGRWT和PGRKO小鼠颗粒细胞的ATAC-seq进一步研究了PGR结合与染色质可及性之间的功能关系。发现在缺乏PGR的情况下,1499个位点的峰值强度显著改变(图5A);有趣的是,在PGRKO中,只有6个位点的ATAC峰值强度增加,而大多数位点在PGRKO中失去了可及性。尽管PGR-染色质结合倾向于颗粒细胞的近端启动子区域,但PGR依赖的ATAC-seq峰值在整个基因组特征中更均匀地分布(图5B)。PGR的消融导致了236个基因的失调,通过对PGRWT和PGRKO颗粒细胞的RNA-seq鉴定出这些基因。一致地,在这些PGR依赖基因的启动子处,ATAC-seq峰值的强度减少是明显的(Spearman相关系数=0.5116,P值<0.0001)(图5C)。这表明这些基因的启动子可及性受到PGR消融的显著影响。以Gpt2为例,其启动子在PGRKO中的可及性不同,在PGR缺失时不显著改变的Abhd2的启动子可及性也显示了PGR依赖的染色质可及性(图5D)。
在PGR驱动的ATAC峰值上进行了转录因子模体富集分析,并与PGR ChIP-seq中富集的模体进行了比较。与ChIP-seq一样,PGR驱动的ATAC峰值序列中最高度富集且最显著的模体是经典的NR3C模体(相对于背景富集了12.4倍)。与PGR ChIP-seq类似,PGR依赖的ATAC峰值中也显著富集了一些非经典模体(图5E)。有趣的是,53%(795个中的1499个)的PGR依赖的ATAC位点被PGR结合,而只有17.8%(267个位点)显示出任何RUNX1结合,其中几乎所有位点(250/267个位点)均被PGR和RUNX1结合(图5F)。此外,PGRKO的ATAC-seq表明PGR通过结合经典NR3C模体直接促进染色质可及性的子集区域。为了验证这一点,我们使用与PGR密切相关的AR HT-SELEX数据生成了NR3C DNA结合的定量模型。从二核苷酸模型派生的能量logo(15个碱基对)符合NR3C共识或PGR模体。接下来,我们使用该模型估计了每个峰值中的PGR结合亲和力,并将其与PGR ChIP-seq峰值强度进行了比较,结果显示PGR结合得分与PGR结合峰值呈现出适度的线性关系。我们通过受试者工作特征(ROC)曲线分析来检查DNA结合模型预测PGR染色质占据的能力,发现结合亲和力对于所有PGR ChIP位点的染色质占据只有适度的预测能力(AUROC = 0.63,n = 13,976),但在也是PGR依赖的ATAC-seq峰值的PGR ChIP峰值上的预测准确性非常高(AUROC = 0.9,n = 804)(图5G)。这得到了强烈的支持,因为与所有PGR结合位点相比,PGR绑定的PGR依赖ATAC峰值在ChIP峰值强度和NR3C模体结合亲和力上表现出明显的增强(图5H)。综上所述,这表明PGR通过结合选定的染色质区域的NR3C模体来驱动染色质重塑,而在非经典位点上的共享PGR/RUNX结合对于促进染色质可及性的依赖性较小。
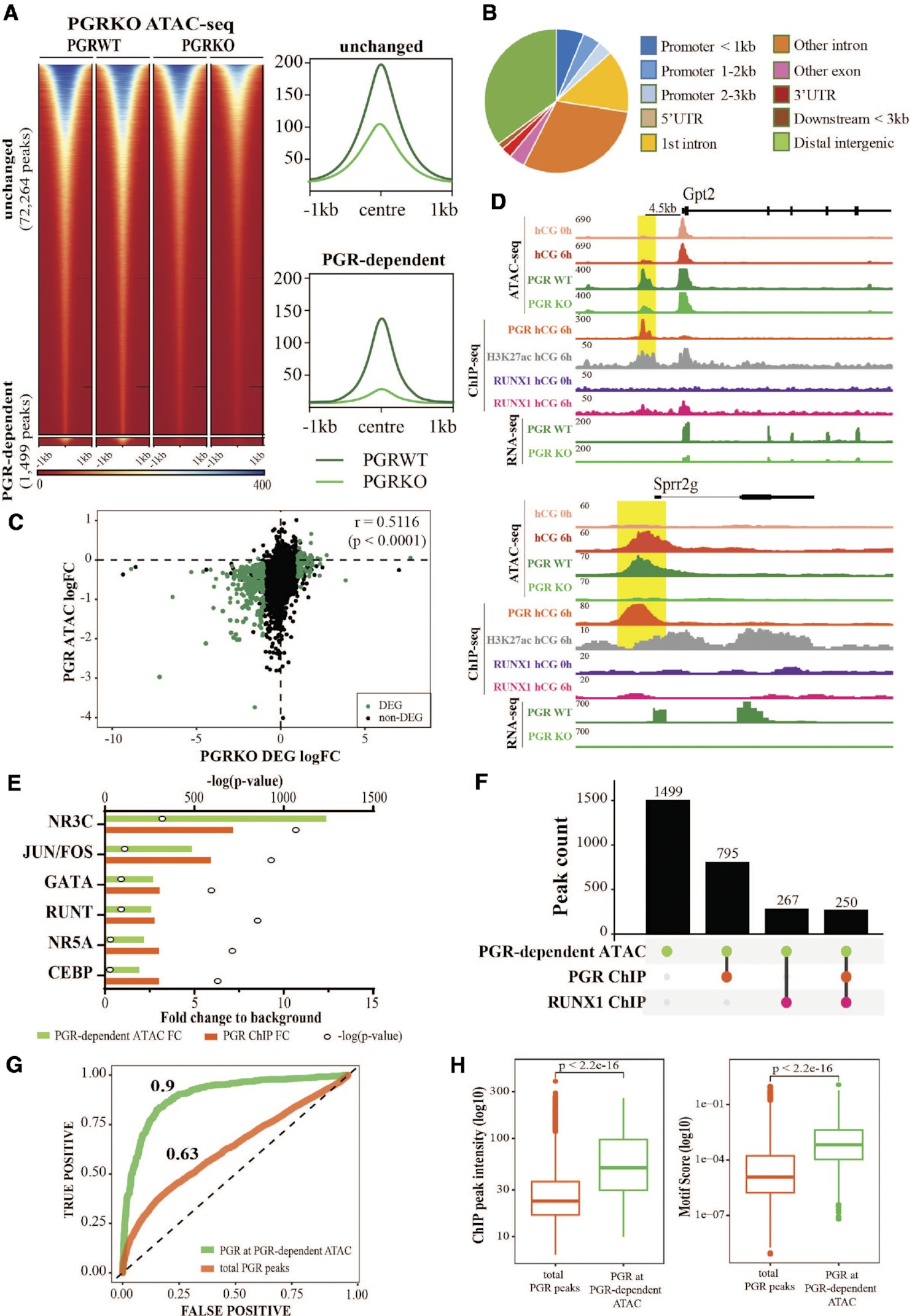
图5.PGR介导颗粒细胞染色质的可及性。
4、PGR亚型介导特异性排卵基因调控
先前的研究已经在总PGRKO和AKO小鼠中记录了受干扰的排卵表型,但在BKO小鼠中没有,通过对这三个小鼠品系的排卵率评估,我们验证了这一点。我们在从每个品系中获取的PGRKO、AKO或BKO和WT同胞中的颗粒细胞中进行了RNA-seq,并通过分层聚类发现,AKO颗粒细胞的全局基因表达模式与总PGRKO相似,而BKO则更接近每个品系的WT(图6A)。在AKO中鉴定了310个基因的差异表达,其中153个(或49.4%)也在缺乏两个PGR亚型的情况下被鉴定出来(图6B)。在这些基因中,48.6%和58.5%的AKO和PGRKO差异表达基因分别也在对LH峰值的响应中差异表达。另一方面,失去PGR-B没有导致显著的转录变化(图6B)。
为了确定共调控下游PGR靶基因的转录因子,使用IPA对DEG数据集的上游调控因子进行了分析(图6C)。如预期,PGR在PGRKO和AKO数据集中被显示为最显著的调控因子。此外,已知被PGR诱导的转录因子,如PPARG和HIF1A,也在两个数据集中富集。重要的是,这种独立的方法还支持RUNX、JUN/FOS和CBP/p300作为PGR依赖性转录反应的介导因子,因为RUNX1、CBFα、JUN、FOS和p300也被通过上游分析鉴定出来。我们在AKO和BKO品系的hCG/孕激素刺激颗粒细胞中进行了PGR/RUNX1和PGR/RUNX2的PLA。在AKO而不是BKO颗粒细胞中观察到与RUNX1和RUNX2的蛋白质相互作用的减少(图6D)。
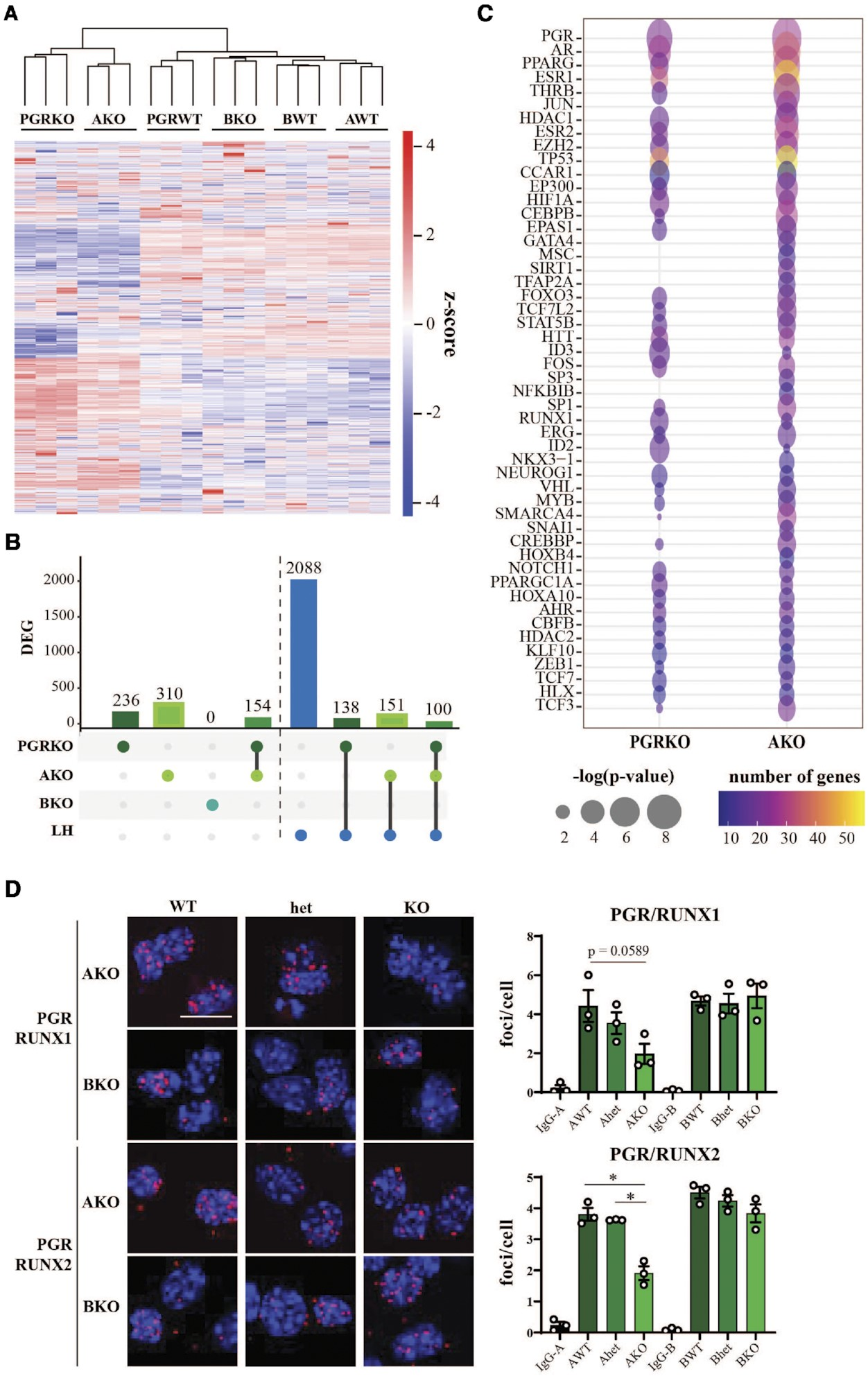
图6.通过与特定转录因子配对的相互作用,围排卵期颗粒细胞中由PGR亚型驱动的转录组。
实验方法
颗粒细胞培养及激素治疗、ATAC-seq、RNA-seq、ChIP-seq、邻位连接试验(PLA)、GFP-TRAP免疫沉淀、质谱。
参考文献
Dinh DT, Breen J, Nicol B, Foot NJ, Bersten DC, Emery A, Smith KM, Wong YY, Barry SC, Yao HHC, Robker RL, Russell DL. Progesterone receptor mediates ovulatory transcription through RUNX transcription factor interactions and chromatin remodelling. Nucleic Acids Res. 2023 Apr 26:gkad271. doi: 10.1093/nar/gkad271. Epub ahead of print. PMID: 37099375.