tsRFun:人类肿瘤tsRNA相关数据库
tsRFun 评估了 32 种癌症的 tsRNA 表达模式和预后价值,并利用高通量 CLASH/CLEAR 或 CLIP 测序数据展示了它们的生物学功能。 总之,tsRFun 为研究 tsRNA 在癌症中的功能和作用提供了有效的数据资源和分析工具。http://rna.sysu.edu.cn/tsRFun/
l Search
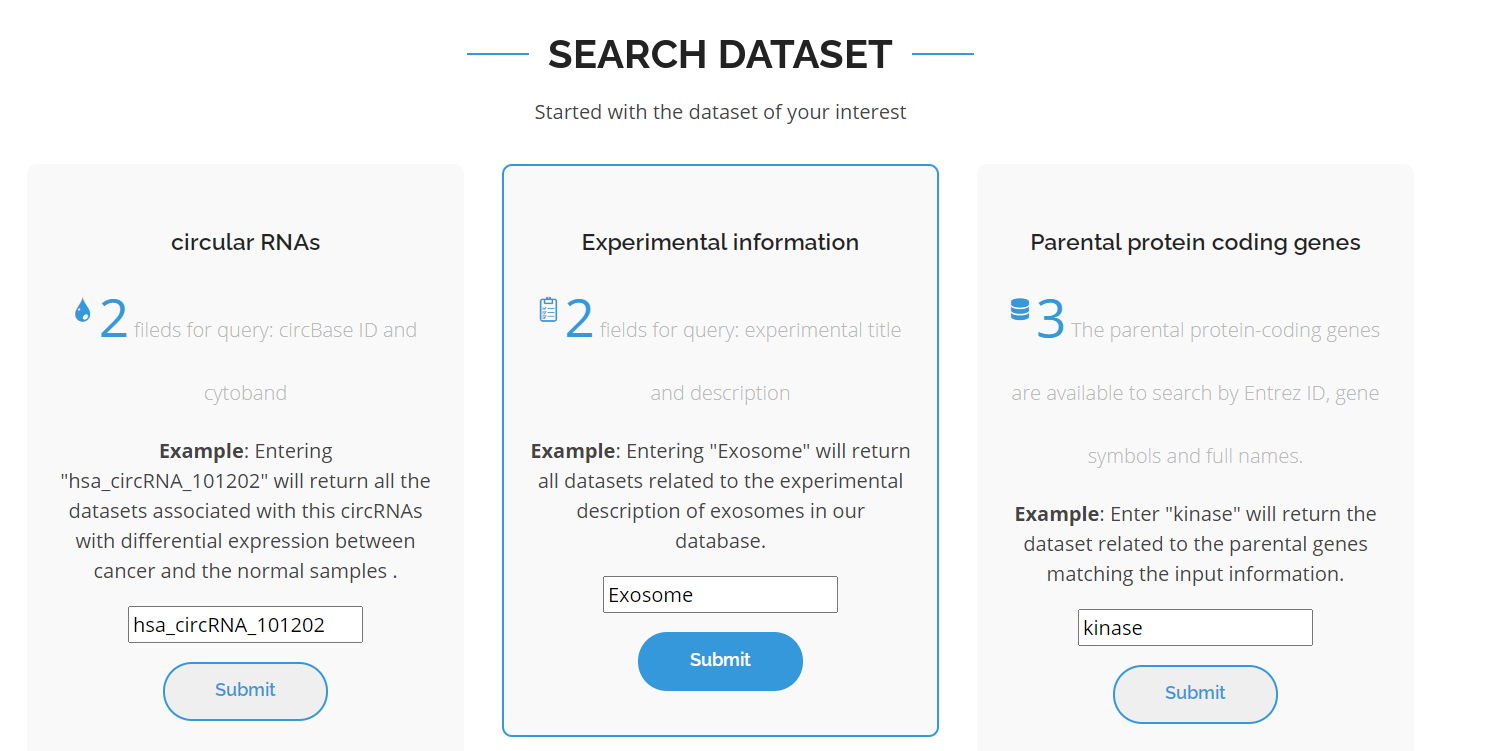
tsRFun 支持多种方式在搜索页面中搜索特定的 tsRNA:
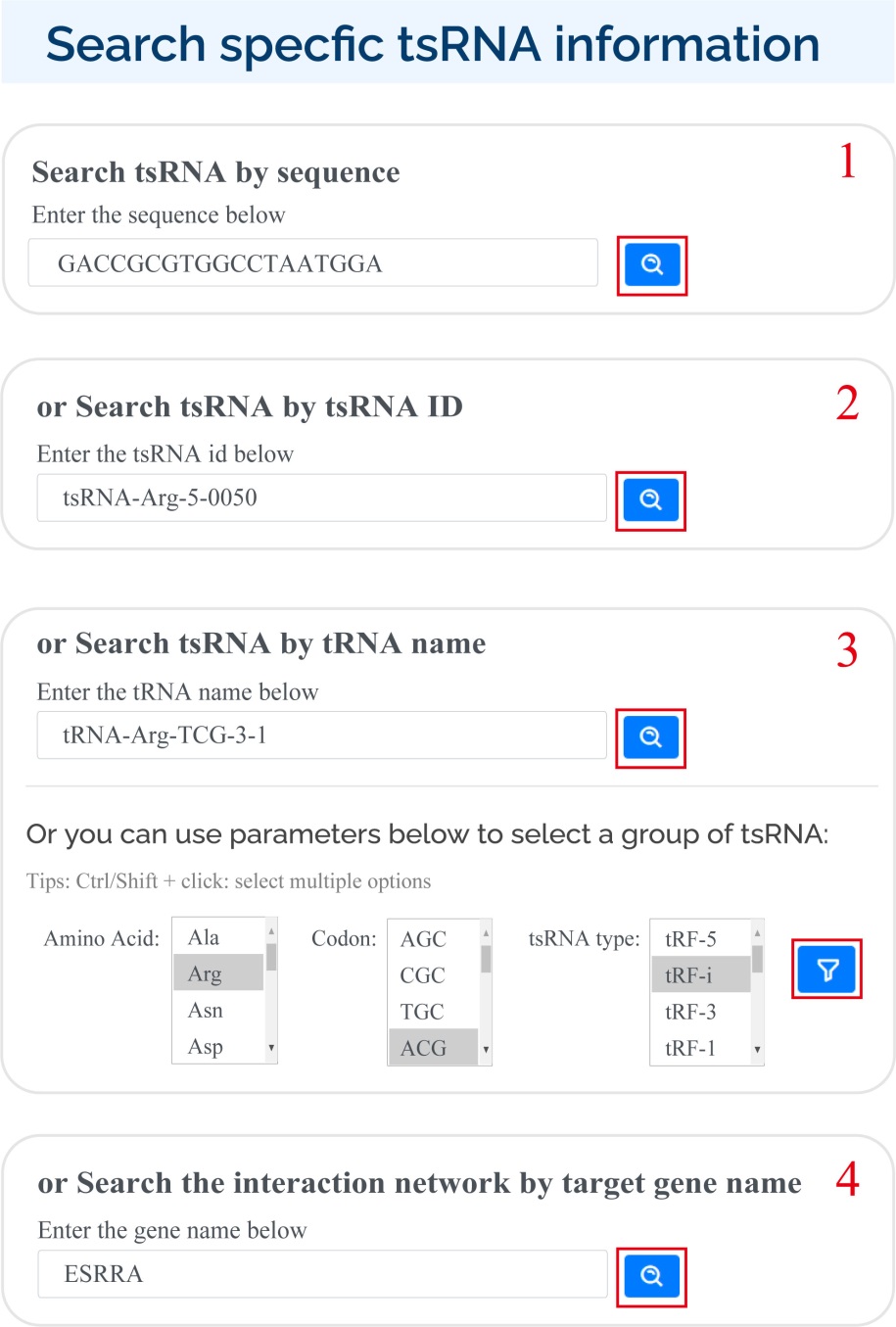
1) 输入序列;
2) 输入tsRNA id;
3) 输入tRNA名称或按氨基酸、tRNA密码子、tsRNA类型选择;4) 输入基因名称。
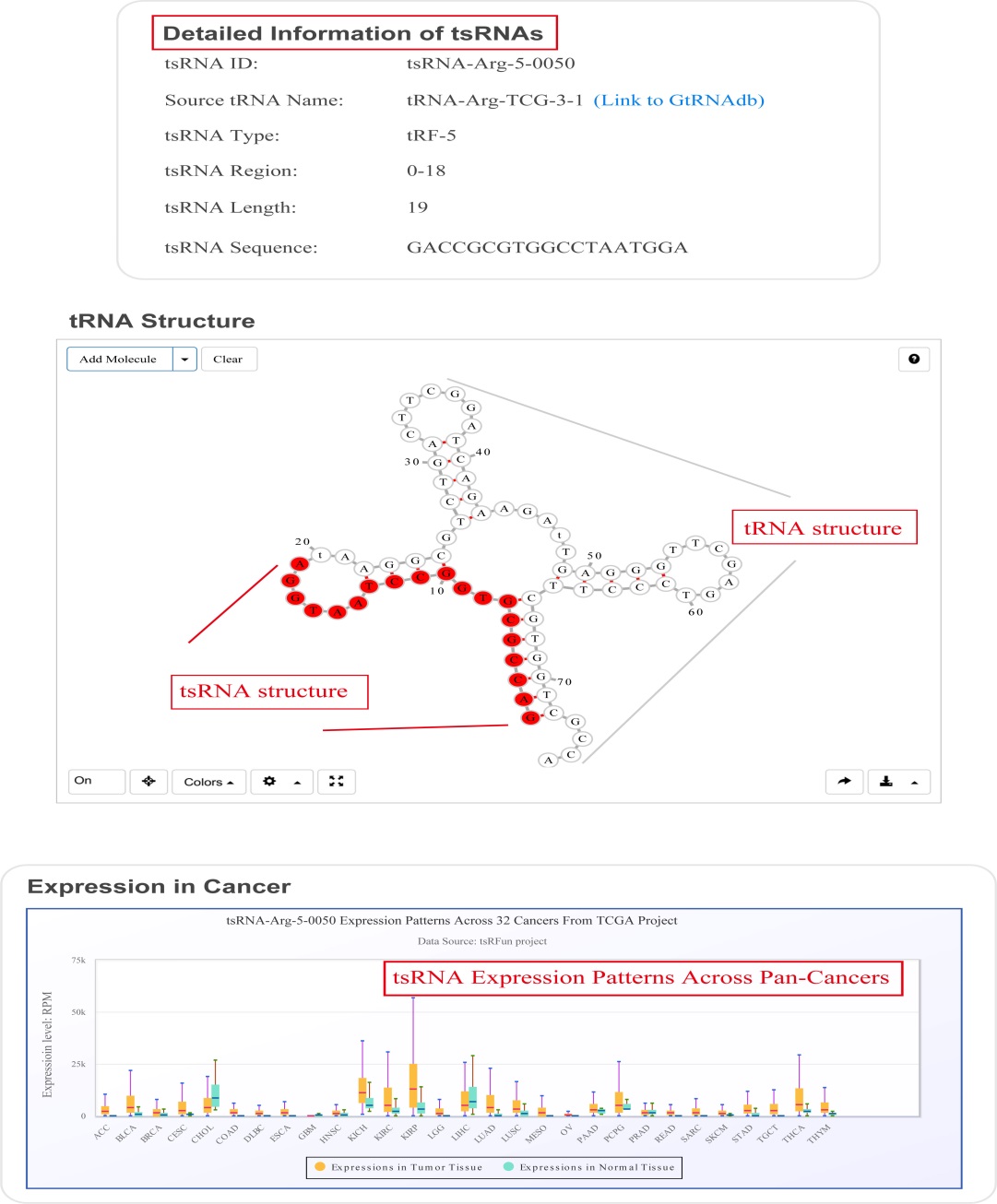
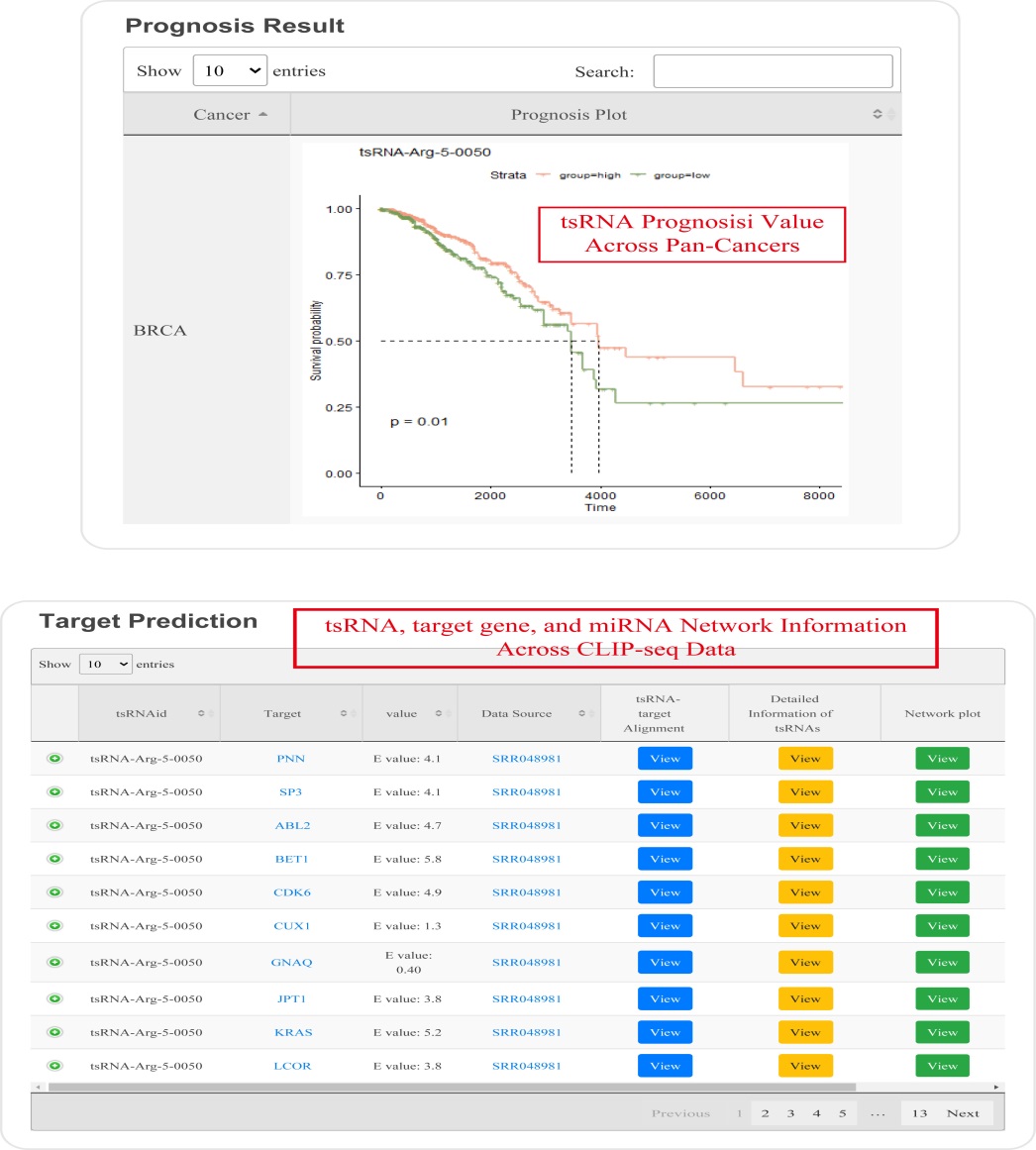
1)或2)的搜索结果为包括二级结构、表达模式在内的tsRNA的详细信息:
l tsRinCancer
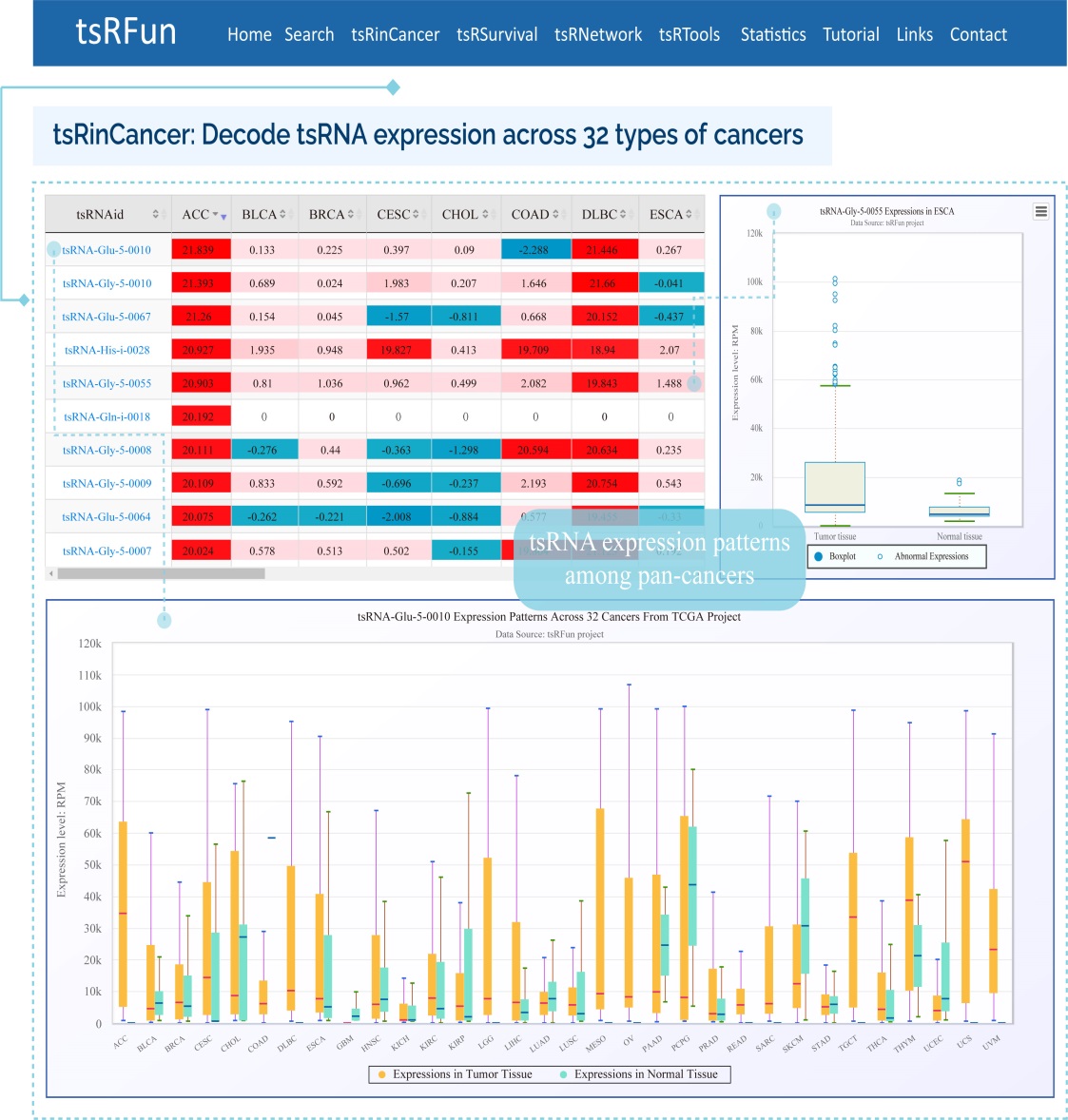
“tsRinCancer”评估 32 种癌症中的 tsRNA 表达模式,将所有样本的平均 RPM 值可视化为箱线图和条形图,并将肿瘤表达相对于正常表达的 log2 倍变化可视化为热图。
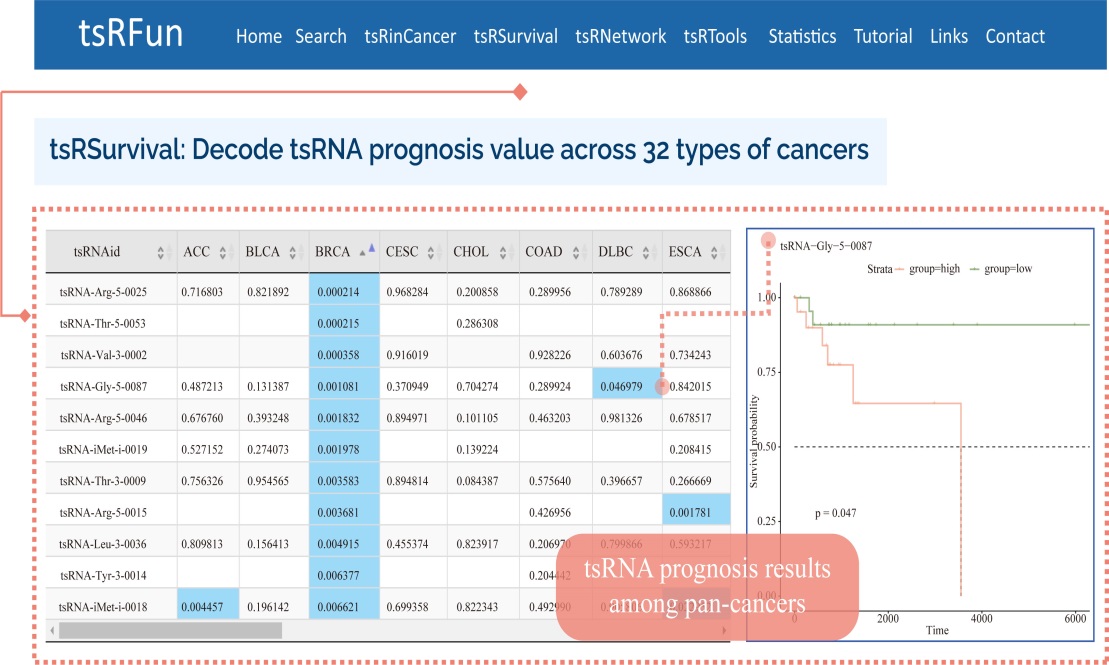
l tsRSurvival
tsRSurvival 通过 Kaplan-Meier 方法和对数秩检验计算 tsRNAs 在不同癌症类型中的预后:
l tsRNetwork
通过超几何测试在tsRNA、microRNA和mRNA之间构建ceRNA网络:

l tsRTools
tsRFun 提供两个实时在线工具tsRFinder 和 tsRTarget,供用户上传自己的序列数据来识别 tsRNA 并预测 tsRNA-target 相互作用。用户需要提交符合要求的数据:fasta 格式

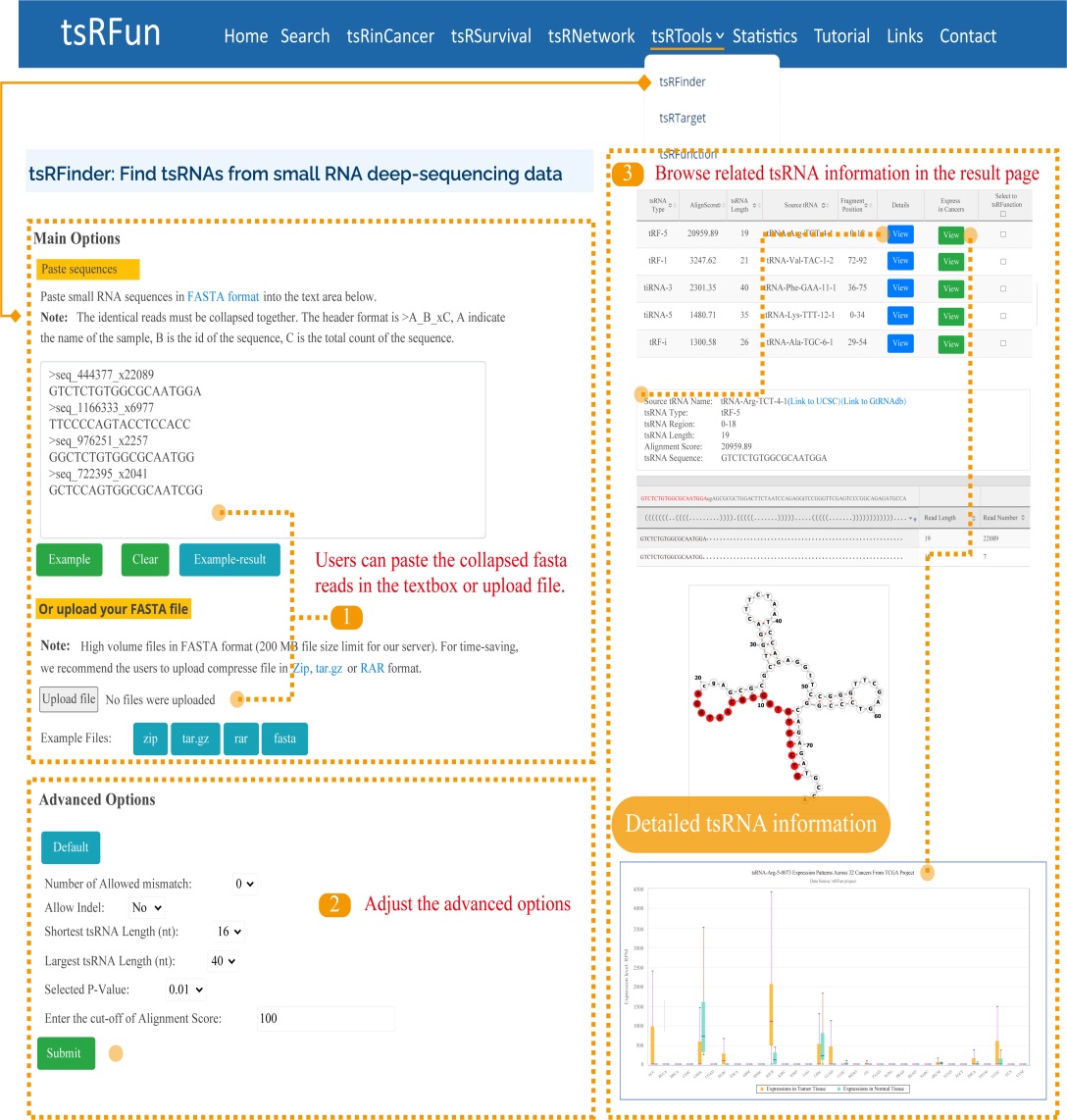
l tsRFinder
“tsRFinder”提供了一种从smallRNA-seq 数据中识别 tsRNA 分子的综合方法:
上传fasta格式的序列信息,选择“Advanced Options”。
i. Number of Allowed mismatch:允许的错配数,允许将smallRNA 读数映射到 tRNA 的最大错配数。 默认为 0,范围为 0 - 2。
ii. Indel:插入缺失,是指核苷酸的插入或缺失。此参数设置允许将small RNA 读数映射到 tRNA 序列的最大核苷酸插入或删除数。
iii. Shortest tsRNA Length (nt):最短 tsRNA 长度 (nt),通常 tRF 的大小为 18 nt,而 tiRNA 的大小为 35 nt。默认为 16,范围为 16 - 40。
iv. Largest tsRNA Length (nt):最大 tsRNA 长度 (nt)”,允许被视为 tsRNA 候选的 tsRNA 的最大长度。 默认为 40,范围为 28 - 40。
v. Selected P-value:选定的 P 值,默认为 0.01。 二项式检验用于评估分布在每个 tRNA 上的已测序 sRNA 缺失的显着性。 在 tRNA 上的连续位置,p 值小于 0.01 的读数被视为 tsRNA 候选。
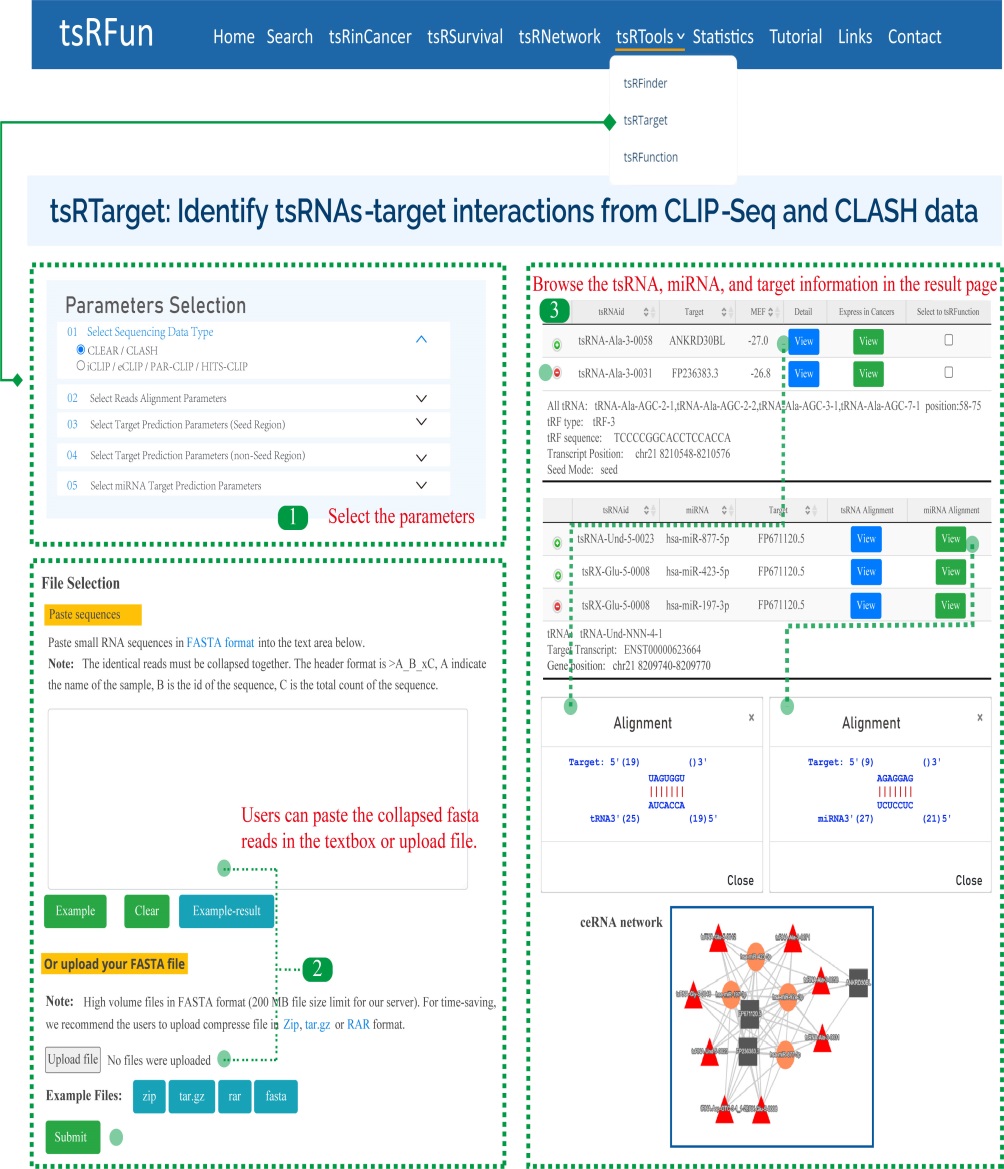
l tsRTarget
“tsRTarget”模块显示了基于 CLIP-Seq 和 CLASH/CLEAR 数据的潜在规范和非规范 tsRNA-mRNA 相互作用。
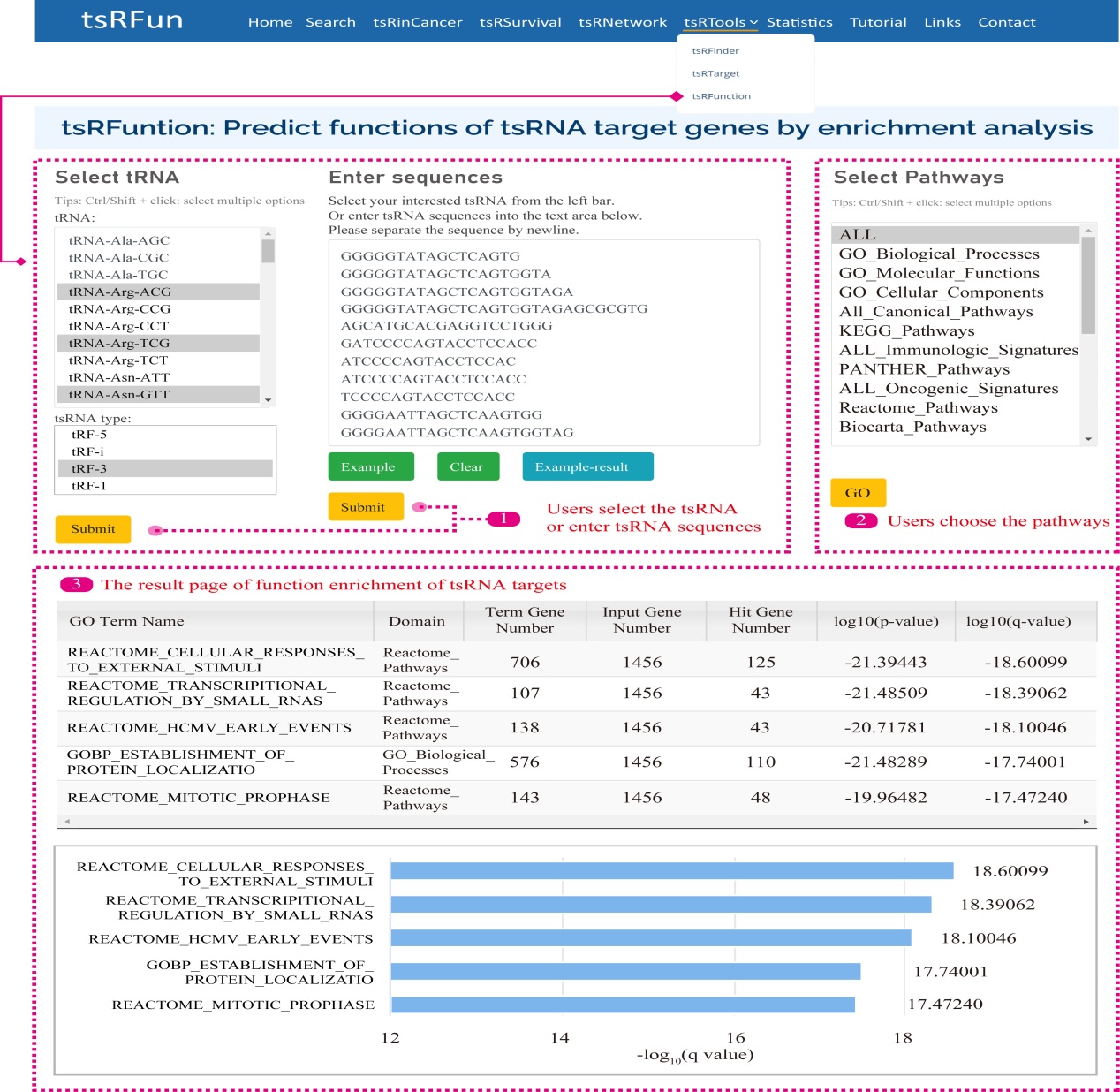
l tsRFunction
“tsRFunction”整合了来自CLASH/CLEAR和CLIP数据预测的tsRNA-target相互作用网络信息,用户可以选择多种tsRNA分子进行后续的功能富集分析。
用户可以通过两种方式查询tsRNAs的功能:(1)选择特定的tsRNA和tsRNA类型; (2) 输入特定的 tsRNA 序列。