EV很无奈!肝脏促炎全靠IRE1A带
内质网至核信号1 (IRE1A)是非酒精性脂肪性肝炎(NASH)患者肝脏中被激活的未折叠蛋白反应的传感器。肝细胞释放的富含神经酰胺的炎性细胞外囊泡(EVs)会激活IRE1A。但是IRE1A激活后对NASH的功能与机制仍不清楚,本文旨在研究抑制IRE1A对饮食诱导的脂肪性肝炎小鼠炎性EVs释放的影响。该文章于2020年6月20发表在Gastroenterology(IF:17.373)期刊上。
技术路线:

主要结果:
1、药物抑制IRE1A可改善NASH
NASH的人和饮食模型可以激活脂毒性内质网应激反应。脂毒性内质网应激激活IRE1A导致EV释放,而胆固醇(FFC)饮食诱发的小鼠脂肪性肝炎与循环促炎EV的增加有关。因此,为了证明IRE1A激活诱导的EV释放和肝脏炎症之间的机制联系,在FFC喂养的小鼠NASH模型中检测了抑制IRE1A对EV释放、肝脏损伤和炎症的影响。如图1A-C所示,FFC饮食引起的剪接Xbp1 mRNA丰度和循环EVs的增加在4µ8C处理的小鼠中显著减弱。不仅如此,4µ8C处理的小鼠中肝炎性病灶,丙氨酸转氨酶 (ALT),巨噬细胞积累都显著减少(图1D-G)。
巨噬细胞介导的炎症在NASH中扮演着重要的作用。募集单核细胞来源的巨噬细胞在NASH中观察到的无菌炎症反应的主要特征。因此,进一步探究抑制IRE1A诱导的EV释放是否影响单核细胞的募集。结果发现,4µ8C处理的小鼠中Cd68、Lgals3、Ly6c (图1H-J)和Ly6c阳性的单核细胞来源巨噬细胞都显著减少。与此一致的是,促炎因子TNF-α和IL1-β(图1L-M)显著降低而抗炎细胞因子IL10 (图1N)保持不变。因此,上述表明抑制IRE1A诱导的EV释放抑制炎症进程。
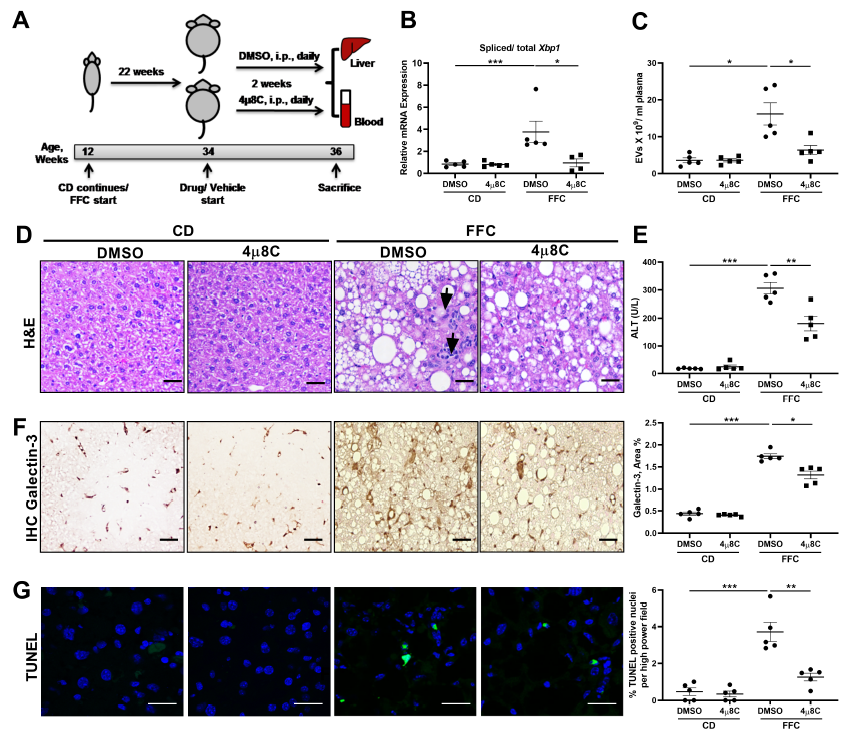
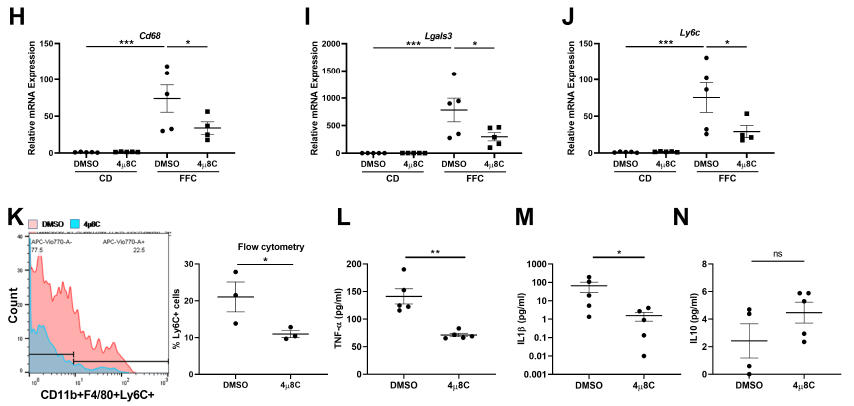
图1 药物抑制IRE1A可抑制NASH
2、基因敲除IRE1A敲除可减弱NASH
作者构建了IRE1A抑制的保肝作用的肝细胞特异性敲除的Ire1a(Ire1aΔhep)小鼠模型。结果发现,Ire1aΔhep小鼠中剪接Xbp1 mRNA丰度、循环中EV的含量、炎性病灶、ALT、巨噬细胞积聚及细胞死亡都显著下降(图2A-G)。巨噬细胞marker Cd68、Lgals3、Ly6c的表达及单核细胞来源的巨噬细胞数量(图2H-I)也在Ire1aΔhep小鼠中显著下降。此外,TNF-α和IL1-β依旧显著降低而IL10也仍保持不变(图2J-L)。因此,在肝细胞IRE1A抑制后NASH减弱与循环EV下降和肝脏单核细胞来源的巨噬细胞减少相关。
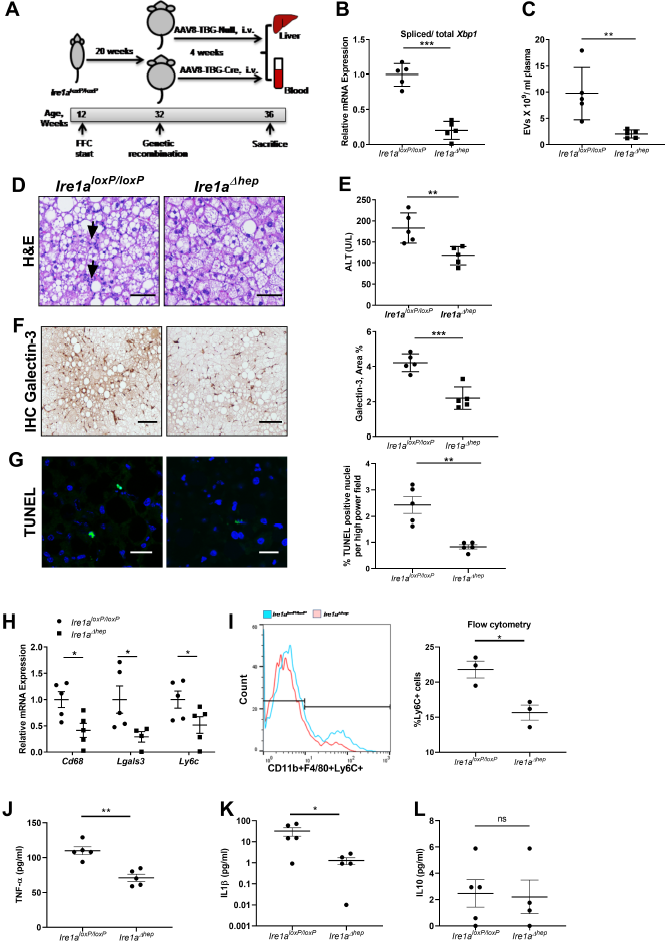
图2 肝细胞特异性IRE1A敲除可减弱NASH
3、IRE1A诱导来源肝细胞EVs
作者接下来探究IRE1A激活是否足以导致肝细胞EV释放。C57BL/6J小鼠接受腺病毒表达IRE1A (AdIRE1A)或B-gal (ads -gal)的对照。结果发现,循环EV在AdIRE1A小鼠中显著高于对照鼠,而这种增长可以被4µ8C减弱(图3A-B)。此外,4µ8C处理显著降低了剪切Xbp1 mRNA丰度(图3C)。IRE1A刺激产生的EV与对照组来源的EV具有不同的大小和形态(图3D)。而具有肝细胞标记物CYP2E1和ASGPR123的免疫电镜进一步证实了循环EV来源于肝细胞(图3E)。使用WB对分离得到的EV进行了表征,随后实验发现无论施用AdIRE1A或者AdB-gal,IRE1A转导的小鼠体内EV的总体数量都会显著增加(图3H)。这种增加是由于肝细胞释放EV导致的。总之,上述表明肝细胞源性EVs的释放在IRE1A激活后显著增加。
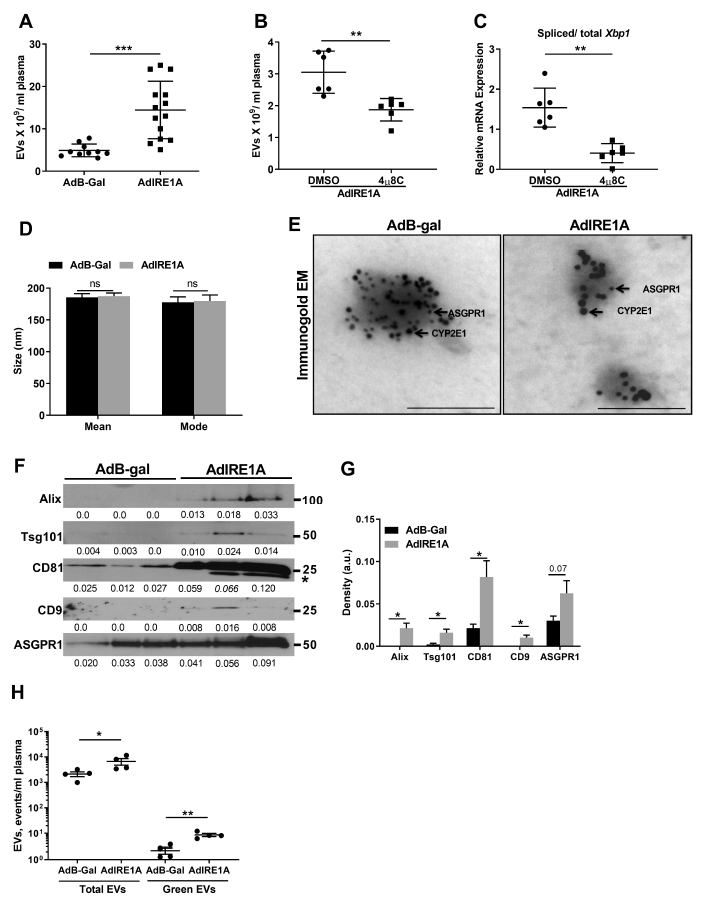
图3 IRE1A诱导肝细胞释放EVs
4、IRE1A刺激的EV吸收单核细胞来源的巨噬细胞进入肝脏
作者接下来检测了CyTOF激活IRE1A后肝内白细胞的细胞异质性,结果发现IRE1A刺激足以导致肝细胞来源的EV释放以及增加单核细胞来源的巨噬细胞在肝脏中的数量。然后,作者分离得到IRE1A刺激EV,并通过尾静脉注射至小鼠体内,结果这些EV几乎全部都能被肝细胞接收(图4A),并且随之巨噬细胞的积累显著增加,这与预期的结果一致(图4B-D)。此外,血浆中细胞因子TNF-α, IL1β, and IL10的水平均没有改变(图4E-G),表明小鼠缺乏系统性炎症,这结果支持EVs在肝微环境中募集单核细胞来源的巨噬细胞的作用。随后,在体外证实IRE1A 刺激的EVs的趋化作用,结果显示BMDM向IRE1A EV的迁移显著增加(图4H-J)。因此,上述数据表明,肝细胞来源的IRE1A EV足以吸引单核细胞来源的巨噬细胞进入肝脏。并且结合以上关于小鼠NASH模型中IRE1A抑制的数据,可以认为IRE1A激活通过刺激吸引单核细胞的EV的释放在NASH中具有促炎作用。
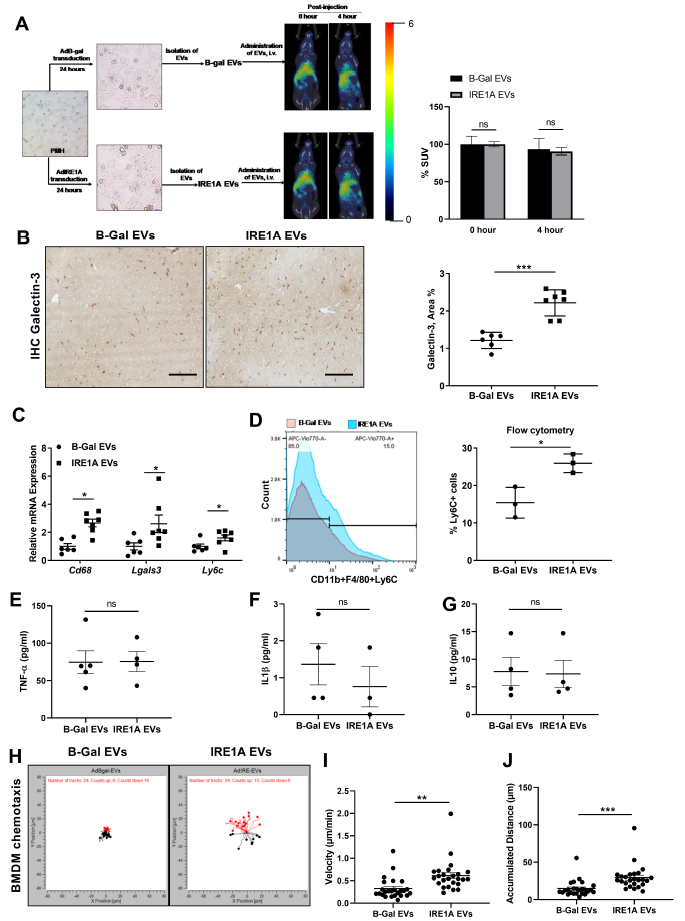
图4 IRE1A刺激的EV吸收单核细胞来源的巨噬细胞进入肝脏
5、IRE1A刺激的EV释放是通过新神经酰胺合成的转录上调介导的
此前的研究发现在分离的肝细胞中,脂毒性EV的释放依赖于神经酰胺的从头合成。因此,作者进一步探究了该通路对体内IRE1A刺激的EV生物合成的作用。如图5A所示,检测到该通路中有8种分子在IRE1A转导的肝脏中富集,其中有6种在IRE1A刺激的EV中富集(图5B)。
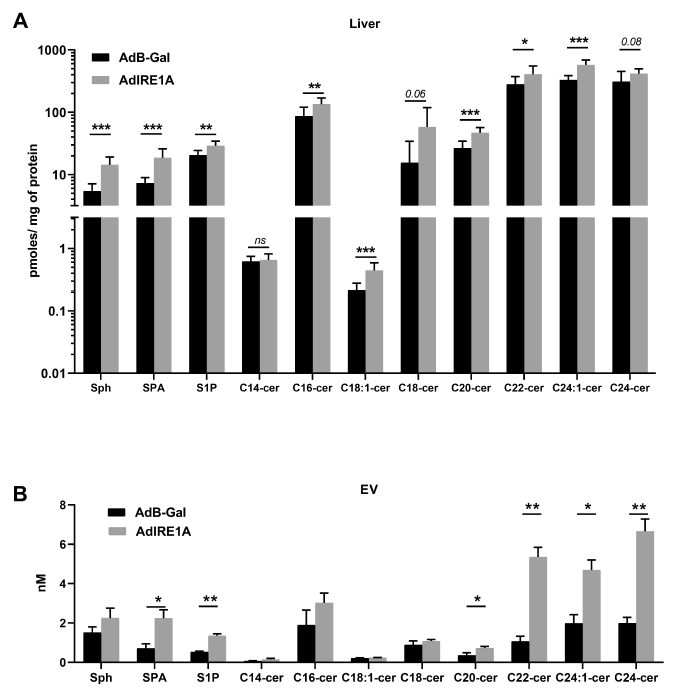
图5 IRE1A诱导神经酰胺新生生物合成
6、从头合成神经酰胺对IRE1A诱导的EV释放是必要的
神经酰胺的合成的限速步骤是SPT催化,为了进一步研究IRE1A对SPT的基因调控,重点研究了XBP1。在silico分析中,确定了在Sptlc1和Sptlc2的启动子区域存在假定的XBP1结合位点(图6A)。IRE1A的激活可诱导Sptlc1和Sptlc2 mRNA和蛋白的表达(图6B,C)。染色质免疫沉淀分析揭示在IRE1A移植的小鼠肝脏中,Sptlc1和Sptlc2启动子上的XBP1占用率显著更高(图6D,E)。ERdj4是一个典型的XBP1靶点,用于指示XBP1的激活(图6F)。因此,作者证实了Sptlc1和Sptlc2在肝脏中的表达增加是一种IRE1A-XBP1依赖的方式并最终导致肝鞘脂类的增加。
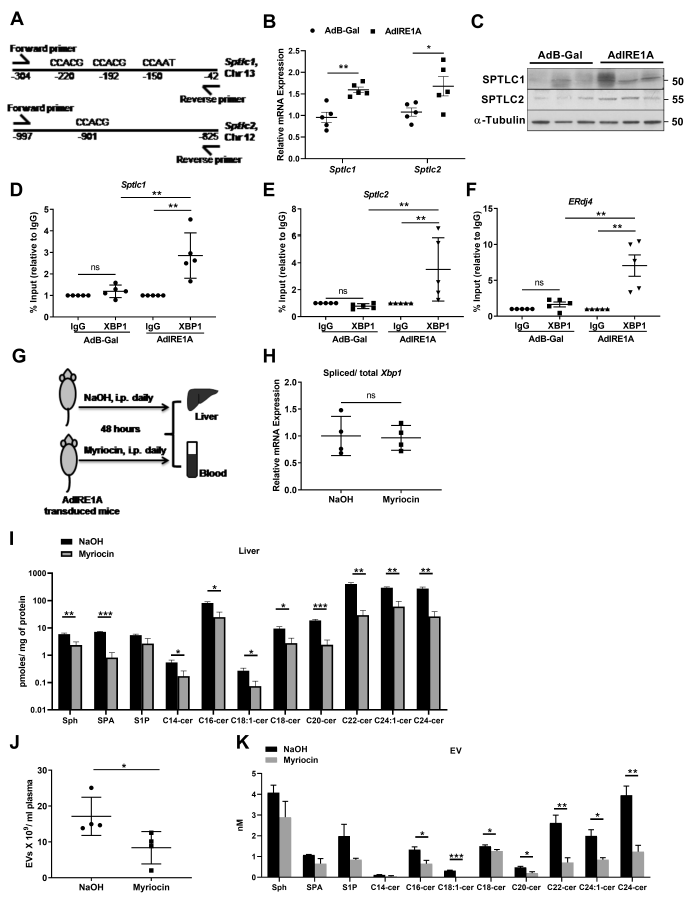
图6 IRE1A刺激的EV释放是通过SPT的转录激活介导的
考虑到IRE1A诱导的肝脏中神经酰胺合成的上调,作者认为抑制神经酰胺合成将阻止EV的释放。通过给小鼠喂食肉豆蔻素,一种SPT的药理学抑制剂(图6G)来探讨这个问题。通过串联质谱可以观察到肉豆蔻素处理小鼠的肝细胞中神经酰胺通路中的分子显著下调,与肉豆蔻素诱导的抑制SPT介导神经酰胺从头合成一致(图6I)。肉豆蔻素处理抑制IRE1A刺激的EV释放,并且EV中神经酰胺的含量也显著下降(图6J-K)。这些结果表明IRE1A刺激的EV释放需要SPT介导的神经酰胺的从头合成。
7、SPTLC1和XBP1丰度升高是人类NASH的特征
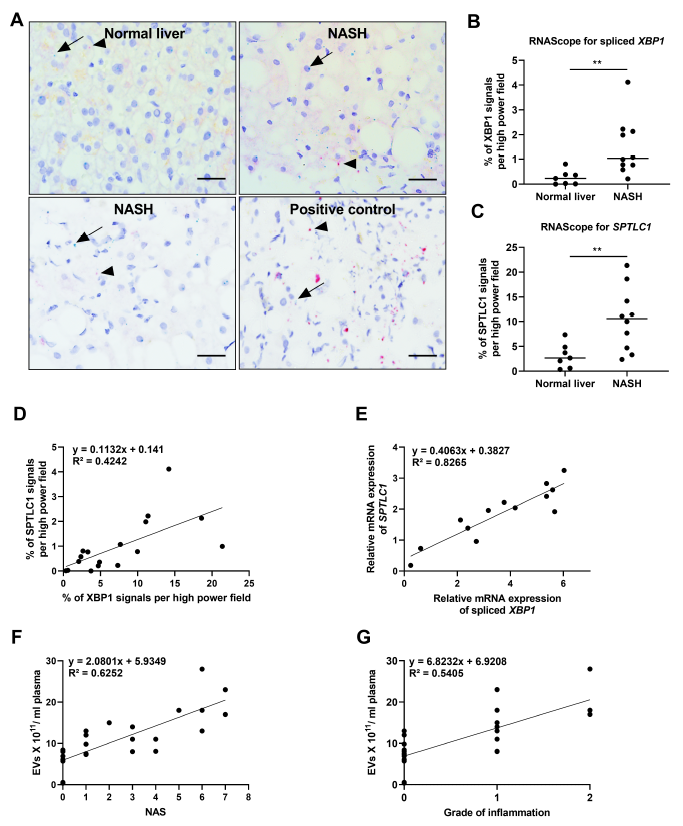
图7人NASH中剪接的XBP1和SPTLC1升高
前面的结果证实了NASH患者循环EV的增加。随后,作者通过与正常对照相比,证明NASH受试者的人类肝脏样本中剪接的XBP1和SPTLC1 mRNA的丰度有所增加(图7),扩展了上述观察结果。使用RNAscope,发现在NASH肝脏中XBP1变异体2 (spliced XBP1)(图7A和B)和SPTLC1(图7A-C)增加。进一步地,spliced XBP1和SPTLC1丰度具有显著的共线性(图7D-E)。最后,将血浆EVs计数与NASH和炎症级别相关联,证明EVs与NASH和炎症均相关(图7F-G)。以上结果表明,IRE1A-XBP1信号通路的激活、XBP1表达的转录靶点SPTLC1及其与NASH和EV计数的相关性,以及EV计数随NASH炎症程度的加重而增加。
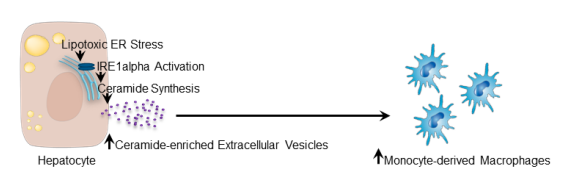
总之,在小鼠肝细胞中,激活的IRE1A通过XBP1促进丝氨酸棕榈基转移酶基因的转录,导致神经酰胺的生物合成和EV的释放。EVs将单核细胞来源的巨噬细胞招募到肝脏,导致饮食诱导脂肪性肝炎小鼠的炎症和损伤。NASH患者肝组织中XBP1、丝氨酸棕榈基转移酶和EV水平均升高。阻断这一途径可能有助于减少NASH患者的肝脏炎症。
参考文献:
Dasgupta Debanjali., Nakao Yasuhiko., Mauer Amy S., Thompson Jill M., Sehrawat Tejasav S., Liao Chieh-Yu., Krishnan Anuradha., Lucien Fabrice., Guo Qianqian., Liu Mengfei., Xue Fei., Fukushima Masanori., Katsumi Tomohiro., Bansal Aditya., Pandey Mukesh K., Maiers Jessica L., DeGrado Timothy., Ibrahim Samar H., Revzin Alexander., Pavelko Kevin D., Barry Michael A., Kaufman Randal J., Malhi Harmeet.(2020). IRE1A Stimulates Hepatocyte-derived Extracellular Vesicles That Promote Inflammation in Mice With Steatohepatitis. Gastroenterology, undefined(undefined), undefined. doi:10.1053/j.gastro.2020.06.031